癌症特异性合成致死基因对预测方法、系统及终端
本发明涉及生物学,特别是涉及一种癌症特异性合成致死基因对预测方法、系统及终端。
背景技术:
1、合成致死(synthetic lethality,sl)是一种基因相互作用类型,其中两个基因的同时突变导致细胞死亡,而单独的一个基因突变则不会产生致命效应。这一策略的理念是,当一个基因具有癌症特异性突变时,扰动另一个基因可以选择性地杀死癌细胞,同时保留健康细胞。这种方法极大地推进了间接瞄准难以治疗的靶点,并拓展了抗癌治疗的靶点范围。该新颖的治疗策略吸引了癌症研究人员的广泛关注,并为肿瘤治疗开辟了新的途径。近年来,基于合成致死性的几种药物已成功进入市场。例如,奥拉帕尼(olaparib)利用了brca1/2基因和parp之间的sl关系,在患有brca1/2突变的卵巢癌患者中取得了突破性的治疗效果。目前,发现sl基因对的现有方法可以分为实验筛选和计算预测。实验筛选通常采用crispr或rnai技术来敲除或破坏目标基因,然后观察癌细胞存活能力的变化。然而,由于人类基因组含有近20,000个基因,且存在数千种不同类型的癌细胞,它们的组合空间太过庞大,难以通过实验筛选来识别新的sl关系。计算方法包括统计推断和基于机器学习的方法。统计方法依赖于从sl概念和已知sl相互作用共享的模式派生出的假设。例如,daisy使用三种假设(即基因组中适者生存的细胞、基于shrna的功能检查和成对基因共表达)来推断sl基因对。尽管统计方法在解释性方面表现优异,但它们严重依赖于从先前知识中推导出的规则,这可能是主观和有偏见的,并且基于不同规则得出的结果可能彼此不一致。机器学习方法则利用已知的sl基因对作为监督学习的标签数据。这些方法可根据其输入数据分为基于知识图谱的方法和基于手工特征的方法。然而,大多数现有的机器学习方法要么单独使用知识图谱,要么单独使用手工特征来表示基因进行sl预测,但往往忽视了两种数据类型的有效整合。
2、尽管一些sl相互作用在不同类型的癌症中是共存的,但许多只存在于少数特定类型的癌症中。最近的研究在三种不同的细胞系中发现了sl基因对,结果显示约10%的sl相互作用仅在两种细胞系中共存,且没有发现在三种细胞系中都存在的sl基因对。尽管用于预测合成致死性的计算方法正在快速发展,但大多数方法都是针对泛癌症sl设计的,并未考虑癌症类型的特异性。并且,大部分方法都需要大量的训练数据上重新训练,然后才能在新类型癌症中预测sl基因对。对于那些具有大量标记的sl基因对用于训练的癌症细胞系,这些方法可能非常有效。然而,对于那些标记稀疏的癌症细胞系,模型可能会倾向于过拟合,从而损害其泛化能力。因此,在少样本情况下进行特定癌症sl的预测仍然是一个具有挑战性的问题。
技术实现思路
1、鉴于以上所述现有技术的缺点,本发明的目的在于提供一种癌症特异性合成致死基因对预测方法、系统及终端,用于解决以上现有技术问题。
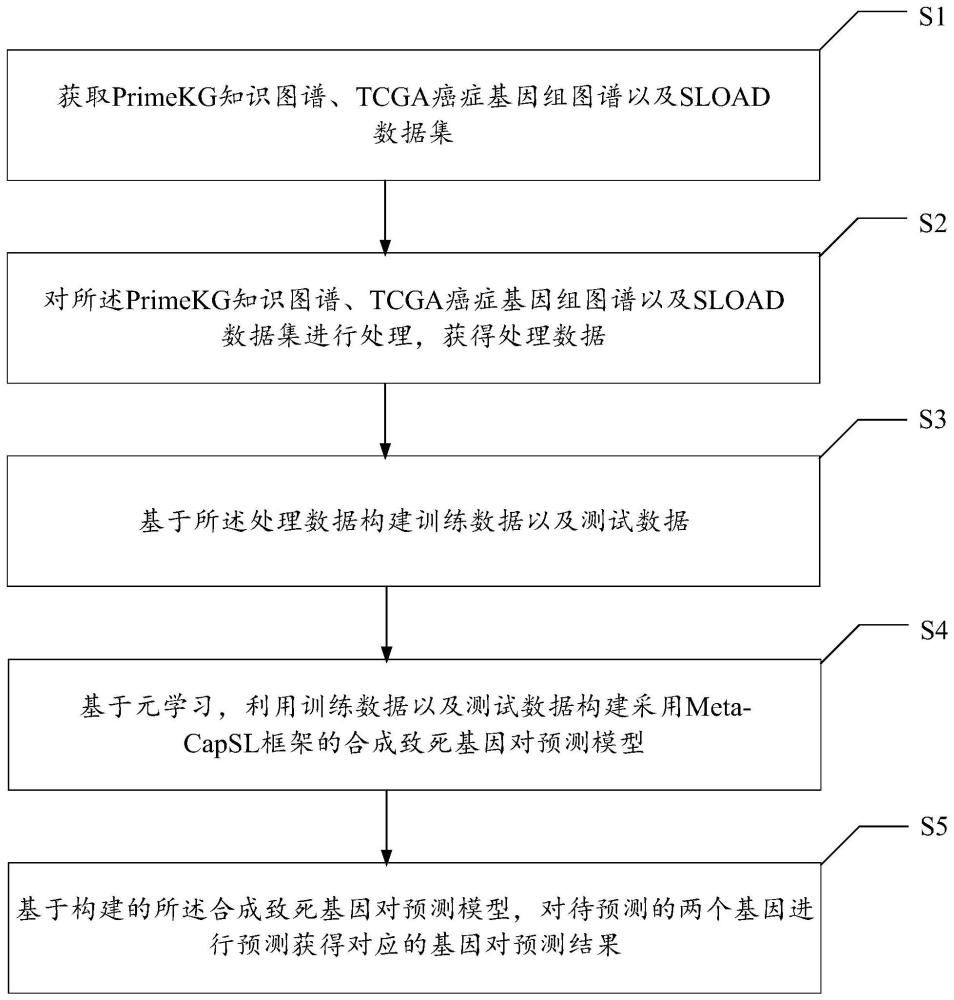
2、为实现上述目的及其他相关目的,本发明提供一种癌症特异性合成致死基因对预测方法,所述方法包括:获取primekg知识图谱、tcga癌症基因组图谱以及sload数据集;对所述primekg知识图谱、tcga癌症基因组图谱以及sload数据集进行处理,获得处理数据;其中,所述处理数据包括:所述primekg知识图谱中各基因对应的知识图谱表征、tcga癌症基因组图谱中各基因对应的癌症特异性基因表达数据以及所述sload数据集中筛选的各目标sl基因对;基于所述处理数据构建训练数据以及测试数据;基于元学习,利用训练数据以及测试数据构建采用meta-capsl框架的合成致死基因对预测模型;基于构建的所述合成致死基因对预测模型,对待预测的两个基因进行预测获得对应的基因对预测结果。
3、于本发明的一实施例中,所述对所述primekg知识图谱、tcga癌症基因组图谱以及sload数据集进行处理,获得处理数据包括:利用边屏蔽预测的自监督任务,根据所述primekg知识图谱获得所述primekg知识图谱中每个基因对应的知识图谱表征;对所述tcga癌症基因组图谱进行预处理,并获得所述tcga癌症基因组图谱中每个基因对应的癌症特异性基因表达数据;筛选sload数据集中各癌症类型对应的湿实验室实验获得的sl基因对,作为各癌症类型的目标sl基因对。
4、于本发明的一实施例中,对所述tcga癌症基因组图谱进行预处理,并获得所述tcga癌症基因组图谱中每个基因对应的癌症特异性基因表达数据包括:对所述tcga癌症基因组图谱中每个基因的fpkm进行对数2转换处理,获得每个基因的预处理fpkm;基于主成分分析法以及每个目标癌症类型的基因共表达网络,根据每个基因的预处理fpkm获得每个基因的癌症特异性基因表达数据。
5、于本发明的一实施例中,所述基于所述处理数据构建训练数据以及测试数据包括:基于所述primekg知识图谱中各基因对应的知识图谱表征、tcga癌症基因组图谱中各基因对应的癌症特异性基因表达数据以及所述sload数据集中筛选的各目标sl基因对生成各癌症类型所对应的多个基因对样本;其中,每个基因对样本包括:基因对中每个基因的知识图谱表征以及癌症特异性基因表达数据;基于各癌症类型的目标sl基因对,对各癌症类型所对应的基因对样本标记sl标签或非sl标签,生成对应各癌症类型的基因对标记样本;筛选sl基因对最多的多个癌症类型,并将筛选的癌症类型的各基因对标记样本作为训练数据;将筛选剩余的目标癌症类型的各基因对标记样本作为测试数据;其中,筛选的目标癌症类型的数量大于筛选剩余的目标癌症类型的数量。
6、于本发明的一实施例中,所述基于元学习,利用训练数据以及测试数据构建采用meta-capsl框架的合成致死基因对预测模型包括:基于模型无关元学习算法maml,利用训练数据以及测试数据对meta-capsl框架进行元训练以及元测试获得合成致死基因对预测模型。
7、于本发明的一实施例中,所述meta-capsl框架包括:多视图特征融合神经网络;其中,所述多视图特征融合神经网络包括:mv编码器,用于将输入的每个基因对的各基因的知识图谱表征以及癌症特异性基因表达数据进行特征融合,获得每个基因对的各基因的融合表征;分类器,连接所述mv编码器,用于基于每个基因对的各基因的融合表征获得对应的基因对致死概率。
8、于本发明的一实施例中,所述mv编码器采用transformer编码器结构,用于将输入的知识图谱表征以及癌症特异性基因表达数据分别投影到它们的潜在特征空间中,并将获得的两个潜向量输入transformer编码器结构进行基于注意力的特征融合,获得融合表征。
9、于本发明的一实施例中,所述基于构建的所述合成致死基因对预测模型,对待预测的两个基因进行预测获得对应的基因对预测结果包括:根据所述primekg知识图谱获得待预测的两个基因的知识图谱表征;对所述tcga癌症基因组图谱中待预测的两个基因的fpkm进行对数2转换处理获得每个基因的预处理fpkm,并获得待预测的两个基因的癌症特异性基因表达数据;基于构建的所述合成致死基因对预测模型,根据输入的待预测的两个基因的知识图谱表征以及癌症特异性基因表达数据获得对应的基因对预测结果。
10、为实现上述目的及其他相关目的,本发明提供一种癌症特异性合成致死基因对预测系统,所述系统包括:数据获取模块,用于获取primekg知识图谱、tcga癌症基因组图谱以及sload数据集;数据处理模块,连接所述数据获取模块,用于对所述primekg知识图谱、tcga癌症基因组图谱以及sload数据集进行处理,获得处理数据;其中,所述处理数据包括:所述primekg知识图谱中各基因对应的知识图谱表征、tcga癌症基因组图谱中各基因对应的癌症特异性基因表达数据以及所述sload数据集中筛选的各目标sl基因对;训练与测试数据获取模块,连接所述数据处理模块,用于基于所述处理数据构建训练数据以及测试数据;模型构建模块,连接所述训练与测试数据获取模块,用于基于元学习,利用训练数据以及测试数据构建采用meta-capsl框架的合成致死基因对预测模型;预测模块,连接所述模型构建模块,用于基于构建的所述合成致死基因对预测模型,对待预测的两个基因进行预测获得对应的基因对预测结果。
11、为实现上述目的及其他相关目的,本发明提供一种癌症特异性合成致死基因对预测终端,包括:一或多个存储器及一或多个处理器;所述一或多个存储器,用于存储计算机程序;所述一或多个处理器,连接所述存储器,用于运行所述计算机程序以执行所述癌症特异性合成致死基因对预测方法。
12、如上所述,本发明是一种癌症特异性合成致死基因对预测方法、系统及终端,具有以下有益效果:本发明通过将每个基因的知识图谱表征以及癌症特异性基因表达数据进行融合,并基于元学习构建采用meta-capsl框架的合成致死基因对预测模型,再通过构建的模型对待预测的两个基因进行预测获得对应的基因对预测结果。本发明用于预测只有少量标记数据的潜在癌症特异性sl基因对,使用了元学习框架来转移不同癌症类型中sl基因对相互作用的元知识,并获得一个能够快速适应新癌症类型的初始化模型。本发明的合成致死基因对预测模型不仅能对基因的知识图谱表征以及癌症特异性基因表达数据进行有效整合提高了预测准确度,还具有良好的性能以及泛化能力。
- 还没有人留言评论。精彩留言会获得点赞!