一种从环境中分离有益微生物的方法与流程
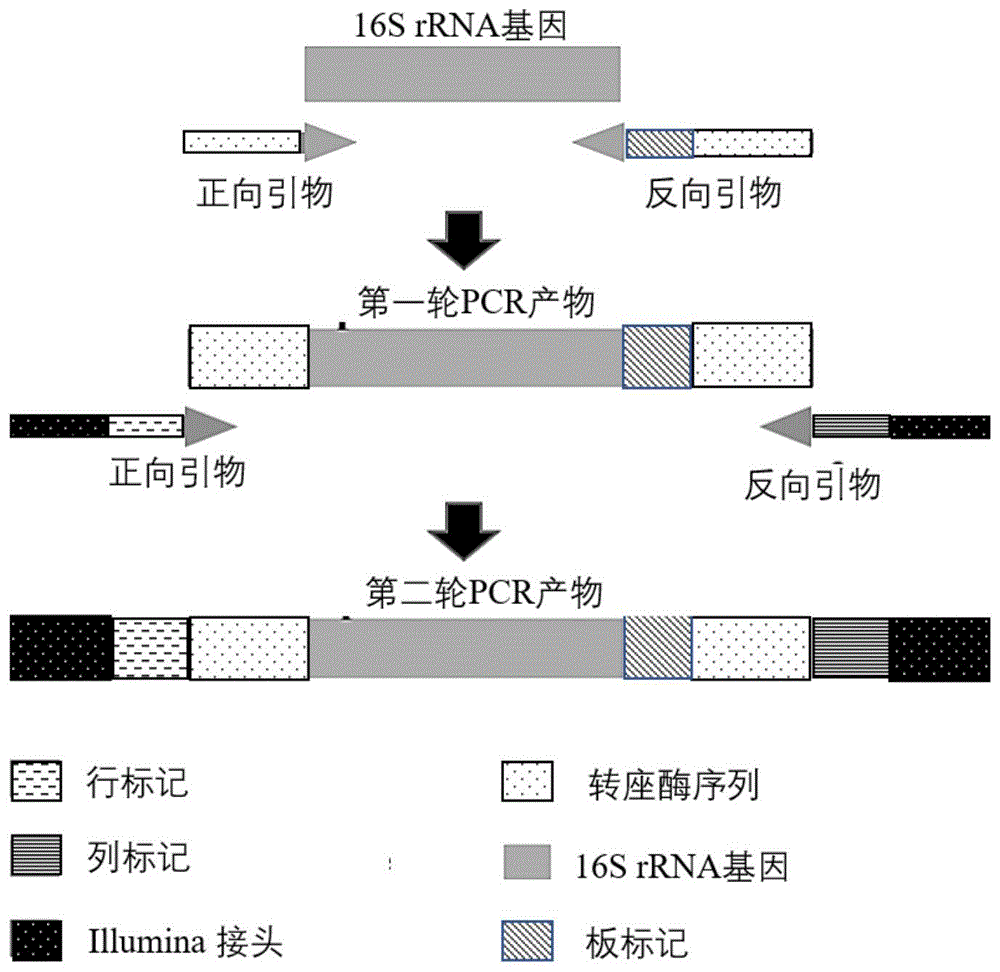
本发明涉及一种从环境中分离有益微生物的方法,特别是从地下害虫为害过的根际土壤中分离活性微生物的方法。
背景技术:
:目前,从环境中筛选有益微生物,主要通过随机采样,从所采样品中分离菌株,然后对所分离的菌株逐一进行活性筛选。这样的筛选过程需要大规模的活性测定,不但耗时耗力,而且效率不高。因此,亟需一种快速挖掘有益微生物的方法。技术实现要素:本发明提供了一种从环境中分离有益微生物的方法,所述有益微生物为对昆虫有杀虫和/或抑制活性的微生物,所述方法包括如下步骤:1)将接种有所述昆虫的环境为第一环境,将不接种所述昆虫的环境为第二环境,2)在所述第一环境中采集第一环境样品,在所述第二环境中采集第二环境样品;3)对所述第一环境样品进行核酸测序,得到第一核酸数据;对所述第二环境样品进行第一环境样品相应类型的核酸测序,得到第二核酸数据;将第一核酸数据与第二核酸数据进行差异性分析,从而从所述第一核酸数据中筛选出不同于所述第二核酸数据的差异性核酸数据;4)对所述第一环境样品中的可培养微生物进行分离培养,得到微生物分离株;5)对所述微生物分离株进行第一环境样品相应类型的核酸测序,得到微生物分离株核酸数据;6)将所述微生物分离株核酸数据与所述差异性核酸数据进行比对分析,找出所述微生物分离株核酸数据与所述差异性核酸数据一致性在96%以上的数据,并将该一致性在96%以上的数据确定为对所述昆虫具有潜在活性的微生物分离株的核酸数据,根据所述潜在活性的微生物分离株的核酸数据找到具有潜在活性的微生物分离株;7)测试所述具有潜在活性的微生物分离株对所述昆虫的杀虫和/或抑制活性。在一个具体实施方式中,所述环境为植物根际土壤。在一个具体实施方式中,所述环境为花生根际土壤。在一个具体实施方式中,在所述昆虫接种到所述环境中1天以上时从所述环境中采集环境样品;优选地,在所述昆虫释放到所述环境中1天至7天是从所述环境中采集环境样品。在一个具体实施方式中,所述昆虫为地下昆虫;所述昆虫为金龟子(scarabaeidae)的幼虫;优选地,所述昆虫为鳃金龟(holotrichia)幼虫;优选地,所述昆虫为暗黑鳃金龟(holotrichiaparallela)幼虫。在一个具体实施方式中,所述核酸测序包括宏基因测序、16s核酸测序和18s核酸测序中的至少一种;优选地,所述核酸测序为16s全长核酸测序和/或16sv3+v4区核酸测序。因此,前文所述相应类型的核酸测序应理解为能进行后续核酸分析的核酸测序类型,比如对所述第一环境样品进行核酸测序的类型为16s全长核酸测序,那么对所述第二环境样品也应进行16s全长核酸测序,或者16sv3+v4区核酸测序,对微生物分离株也应进行16s全长核酸测序,或者16sv3+v4区核酸测序;反过来,对所述第一环境样品进行核酸测序的类型别为16sv3+v4区核酸测序,那么对所述第二环境样品也应进行16s全长核酸测序,或者16sv3+v4区核酸测序,对微生物分离株也应进行16s全长核酸测序,或者16sv3+v4区核酸测序。这样才能对两种环境样品以及微生物分离株的核酸序列数据进行分析。在一个具体实施方式中,当所述第一环境样品为多个时,对所述多个第一环境样品分别进行核酸测序,得到多个第一核酸数据;或对所述多个第一环境样品的核酸分别进行第一样品标记后,混合所述多个第一环境样品标记后的核酸,进行核酸测序,将测序后的数据根据第一样品标记区分开所述多个第一环境样品的核酸数据,得到多个第一核酸数据。在一个具体实施方式中,当所述第二环境样品为多个时,对所述多个第二环境样品分别进行核酸测序,得到多个第二核酸数据;或对所述多个第二环境样品的核酸分别进行第二样品标记后,混合所述多个第二环境样品标记后的核酸,进行核酸测序,将测序后的数据根据第二样品标记区分开所述多个第二环境样品的核酸数据,得到多个第二核酸数据。在一个具体实施方式中,当所述微生物分离株为多个时,对所述多个微生物分离株分别进行核酸测序,得到多个微生物分离株核酸数据;或对所述多个微生物分离株的核酸分别进行微生物分离株样品标记后,混合所述多个微生物分离株标记后的核酸,进行核酸测序,将测序后的数据根据微生物分离株样品标记区分开所述多个微生物分离株的核酸数据,得到多个微生物分离株核酸数据。在一个具体实施方式中,所述微生物分离株样品标记包括板标记、行标记和列标记。在一个具体实施方式中,所述第一核酸数据和所述第二核酸数据独立地为相对丰度大于等于0.0005的otus;所述差异性分析为随即森分析,所述差异性核酸数据为差异性otus;所述比对为本地blast,所述潜在活性分离株的核酸数据为潜在活性分离株的otus。在一个具体实施方式中,所述差异性核酸数据为选取位于前30位的所述差异性otus所定义的差异性关键otus,以用于与所述微生物分离株核酸数据进行比对分析。在一个具体实施方式中,所述差异性核酸数据为选取位于前20位的所述差异性otus所定义的差异性关键otus,以用于与所述微生物分离株核酸数据进行比对分析。在一个具体实施方式中,所述差异性核酸数据为选取位于前15位的所述差异性otus所定义的差异性关键otus,以用于与所述微生物分离株核酸数据进行比对分析。在一个具体实施方式中,所述差异性核酸数据为选取位于前10位的所述差异性otus所定义的差异性关键otus,以用于与所述微生物分离株核酸数据进行比对分析。在一个具体实施方式中,所述差异性核酸数据为选取位于前5位的所述差异性otus所定义的差异性关键otus,以用于与所述微生物分离株核酸数据进行比对分析。在一个具体实施方式中,所述第一核酸数据、所述第二核酸数据和所述微生物分离株核酸数据为独立地在96%以上的水平下进行聚类的otus。在一个具体实施方式中,所述第一核酸数据、所述第二核酸数据和所述微生物分离株核酸数据为独立地在97%以上的水平下进行聚类的otus。在一个具体实施方式中,在对所述第一核酸数据、所述第二核酸数据和所述微生物分离株进行otus聚类之前,根据本领域的常规方法对测序数据去除引物和/或标记等处理,进行必要的拼接,以及去除嵌合体得到各自的有效数据。在一个具体实施方式中,利用rdp方法和greengene数据库对为otus的所述第一核酸数据、所述第二核酸数据和所述微生物分离株核酸数据独立地在0.8至1的阈值下进行物种注释分析。本发明的有益效果:本发明首次发现在昆虫对植物的地下部分造成为害后,能够改变植物根际微生物群落菌落结构,特别是对目标害虫具有杀虫活性的关键微生物种类的改变。结合16srrna基因高通量测序和培养组学方法,利用随机森分析寻找代表菌种otus,然后对代表性菌株进行生测。与传统方法对所有分离菌株进行大规模生测相比,大大降低了生测的规模和劳动量,节省了时间,而且通过该方法筛选到活性菌株的几率有了明显的提升。最为有利的是,该方法并不仅仅限于对目前熟知的例如苏云金芽胞杆菌和荧光假单胞菌等物种的新菌株的筛选,而是可以扩大到所有可能的潜在物种,因此,更有利于发掘新的具有杀虫活性的物种或菌株类型。此外,由于通过该方法筛选得到的菌株本身来自于植物根际,因此将其用于植物根际比来自于虫尸等环境的菌株将具有与植物更好的相容性和定殖能力等优势。附图说明图1为使用带有板标记、行标记和列标记的特异引物的两轮pcr示意图。图2显示了随机森来分析出蛴螬处理和健康花生对照的花生根际细菌群落在1d时存在差异性otus,其中,横坐标meandecreasegini为分类减少准确度。图3显示了随机森来分析出蛴螬处理和健康花生对照的花生根际细菌群落在2d时存在差异性otus,其中,横坐标meandecreasegini为分类减少准确度。图4显示了随机森来分析出蛴螬处理和健康花生对照的花生根际细菌群落在7d时存在差异性otu,其中,横坐标meandecreasegini为分类减少准确度s。具体实施方式以下通过优选的实施案例的形式对本发明的上述内容作进一步的详细说明,但其不构成对本发明的限制。如无特别说明,本发明的实施例中的试剂均可通过商业途径购买。以下实施例中所用的暗黑鳃金龟(holotrichiaparallela)由河北省沧州市农林科学院植物保护研究所提供;所种植的花生(arachishypogaealinn.)品种由山东花生研究所提供:花育22号。16srrna基因二代测序工作由北京百迈客生物科技有限公司完成,16srrna基因二代测序数据分析由北京赛默百合生物科技有限公司完成。实施例11.1田间花生种植在河北廊坊实验基地试验田种植花生,在播种前在田间深埋足够数量的304不锈钢网箱以限制后续接种的蛴螬向周边扩散,网箱的位置为1亩地的四角和中间点,共5个点,每个点深埋2个网箱,分别用于蛴螬处理和健康花生对照。每个304不锈钢网箱中种有九棵花生。每3天浇一次水。1.1.1蛴螬处理到花生结荚期,向304不锈钢网箱中的花生周围接种已经饥饿处理48h的暗黑鳃金龟三龄幼虫,每棵花生植株周围接入4头。分别在接种暗黑鳃金龟幼虫后的1d、2d和7d时,每个时间点从304不锈钢网箱采用五点法采集花生根际土。每棵花生为1个重复,每个304不锈钢网箱点3个重复。五点采样中的每个重复的根际土不混合,每个时间点以及不同时间点采集的花生根际土也不混合。即一颗花生的根际土壤构成一个样品。如果所采集的花生根系发生了病害,则不将其计入样品内,也不再新增重复,最终将缺少一个样品。最终1d时间点采集到15个样品,2d时间点采集到15个样品,7d时间点采集到11个样品。样品数量总计41个。1.1.2健康花生对照以不接种暗黑鳃金龟幼虫的健康花生作为对照,与蛴螬处理同时平行进行。最终1d时间点采集到12个样品,2d时间点采集到11个样品,7d时间点采集到11个样品。样品数量总计34个。1.2田间根际土壤采集及处理每颗花生根际土采集及处理如下:完整取出每棵花生的根系,抖落下花生根上的非根际土,仅留下与根表距离<1mm的根际土,用无菌剪刀剪下带有根际土的花生根系,将带有根际土的花生根系放至无菌的50ml离心管中,加入1×pbs缓冲液,直至没过整个花生根系,浸泡静置15min后,涡旋震荡15s,将花生根系取出后,用100目筛过滤掉根际土壤悬液中的须根、杂质等,然后离心3200g,15min,在去掉部分上清后,用留下的5ml上清重新悬浮离心沉淀物(即根际土),得到每棵花生的根际土壤悬液,共获得75个样品的根际土壤悬液。将每棵花生的根际土壤悬液分为两部分,其中2ml(约0.25g根际土)用于提取土壤细菌总基因组;剩余的加入50%体积的甘油保存,以用于后期根际细菌的分离。1.3单个样品的根际土壤细菌总基因组的提取单个样品的根际土壤细菌总基因组的提取按照土壤细菌总dna提取试剂盒(dnaisolationkit)说明书进行提取,对破碎步骤稍作改动:将powerbead中的液体和磁珠转移到装有100mg土壤样品的管中,摇匀后加入60μl试剂盒中提供的溶液c1后涡旋震荡,放入组织破碎仪中的tissuelyserltadapter适配器中,30hz,破碎45秒(s),停10s,总共破碎1min30s。所提取的单个样品的根际土壤细菌总基因组通过0.7wt%琼脂糖凝胶电泳检测质量,然后于-20℃保存备用。实施例2根际土壤细菌16srrna基因v3+v4区测序及分析由北京百迈客生物科技有限公司以提取的单个样品的根际土壤细菌总基因组为模板,使用16srrna基因v3+v4区的通用引物341f(seqidno.1)和806r(seqidno.2)进行pcr扩增,pcr产物纯化,然后借助于建库试剂盒(dnapcr-freesamplepreparationkit),通过pcr给每个样品加上样品标记(barcode),并最终构建文库,用qubit和q-pcr定量检测所建文库的质量,然后在hiseq2500pe250平台上进行二代测序。将得到的最原始的数据根据样品标记区分75个样品的数据,并去掉标记和通用引物序列信息,得到75个样品的16srrna基因v3+v4区测序数据。将蛴螬处理和健康花生对照总计获得的75个样品的16srrna基因v3+v4区测序数据的读长(reads)分别使用flash(v1.2.7)(tanjam,salzbergsl.flash:fastlengthadjustmentofshortreadstoimprovegenomeassemblies.bioinformatics,2011,27(21):2957-2963)拼接后获得v3+v4区的原始数据(rawtags)。原始数据经过质量过滤(trimmomatic[bolgeram,lohsem,usadelb:trimmomatic:aflexibletrimmerforilluminasequencedata.bioinformatics2014:btu170],version0.33)处理后得到cleantags,其中参数为设置:50bp的窗口,如果窗口内的平均质量值低于20,从窗口开始截去后端碱基,过滤质控后长度小于tags长度75%的tags,得到高质量的tags数据(cleantags)。使用uchime(edgarrc,haasbj,clementejc,quincec,knightr:uchimeimprovessensitivityandspeedofchimeradetection.bioinformatics2011,27(16):2194-2200)将cleantags与golddatabase数据库进行比对以去掉cleantags中的嵌合体序列(haasbj,geversd,earlam,etal.chimeric16srrnasequenceformationanddetectioninsangerand454-pyrosequencedpcramplicons.genomeresearch,2011,21(3):494-504.),最后得到75个样品的有效数据(effectivetags)。将蛴螬处理和健康花生对照总计获得的75个样品的有效数据分别通过uparse软件(uparsev7.0.1001)(edgar,2013)进行otu聚类,以一致性在97%以上作为标准将序列聚类成otus。利用rdpclassifier(version2.2)(wangq,garritygm,tiedjejm,etal.naivebayesianclassifierforrapidassignmentofrrnasequencesintothenewbacterialtaxonomy.applied&environmentalmicrobiology,2007,73(16):5261-5267.)方法和greengene数据库对otus进行物种注释分析(阈值设定为0.8~1)(desantistz,hugenholtzp,larsenn,etal.greengenes:chimera-checked16srrnagenedatabaseandworkbenchcompatibleinarb.applied&environmentalmicrobiology,2006,72(7):5069-5072.)。筛选出相对丰度大于等于0.0005的otus,以用于后续分析。在百迈客云平台微生物多样性分析平台上采用随机森来分析出蛴螬处理和健康花生对照的花生根际细菌群落在1d、2d和7d时分别存在的差异性otus。结果见图1至图3。图1至图3分别显示了导致群落组成差异的前30个otus。其中,将位于前5位的otus定义为差异性关键otus,蛴螬处理和健康花生对照之间1d时的差异性关键otus为:5735、503、26、53和124;2d时的差异性关键otus为6162、2338、1924、4657和471;7d时的差异性关键otus为30、1154、4657、137和355。基于该分析,得到如上所列15个差异性关键otus。实施例33.1根际可培养细菌的分离tsb培养基:酪蛋白水解物17.0g,大豆蛋白胨3.0g,nacl5.0g,k2hpo42.5g,葡萄糖2.5g,用蒸馏水定容至1.0l。tyg培养基:胰蛋白胨1.0g,酵母提取物1.0g,葡萄糖0.5g,kcl6.34g,nacl1.2g,mgso4·7h2o0.25g,k2hpo40.13g,cacl2·2h2o0.22g,k2so40.17g,na2so42.4g,nahco30.5g,na2co30.09g,fe·edta0.07g,用蒸馏水定容至1.0l。其中,cacl2·2h2o、nahco3和葡萄糖采用过滤除菌。m408培养基:酵母提取物1.0g,甘露醇10.0g,k2hpo40.5g,mgso4·7h2o0.2g,nacl0.1g,用蒸馏水定容至1.0l。其中,甘露醇采用过滤除菌再添加到灭菌后的其他组分中。yem培养基:酵母提取物0.5g,甘露醇5.0g,k2hpo40.5g,mgso4·7h2o0.2g,nacl0.1g,用蒸馏水定容至1.0l。其中,甘露醇采用过滤除菌再添加到灭菌后的其他组分中。面粉培养基:普通面粉4.0g。twye培养基:酵母提取物0.25g,k2hpo40.5g,矿泉水定容到1.0l。lb培养基:酵母提取物5g,胰蛋白胨10.0g,nacl10.0g,用蒸馏水定容至1.0l。除特别说明之外,以上液体培养基均在121℃高压蒸汽灭菌20min。固体培养基则是在以上液体培养基中按1.5%的比例加入琼脂粉,121℃高压蒸汽灭菌20min,冷却到适温倒入9cm培养皿,制备固体平板。参照bai等人的文章(baietal.,2015),使用tyg、tsb、twye、yem、m408和面粉固体培养基平板对根际土壤悬液中的可培养细菌进行分离:首先将41个蛴螬处理的加有50%体积甘油的根际土壤悬液样品混合在一起,然后进行10倍梯度稀释,选取稀释103、104、105三个梯度进行涂板,每个重复涂40μl土壤悬液,于30℃恒温培养箱中培养1周,1个平板计作1个重复,每个梯度3个重复。一周后,发现稀释至103时,每个平板上生长的菌落量较大,且十分密集,难以挑取单菌落进行再次培养;稀释至105时,每个平板上生长的菌落过少;只有稀释至104时,每个平板上生长的菌落数量在100-300个之间,因此选用该稀释梯度挑取菌落。将挑取的菌落于96孔板中在与平板一致的液体培养基中培养,其中,每孔600μl培养基,共获得43板96孔板的分离株,共计4128孔分离株(由于没有进行纯化培养,因此每个孔中的分离株不视为单克隆菌株)。于30℃摇床中培养,直至菌液浑浊。每孔吸出200μl菌液,将吸出4128孔分离株的菌液混合,作为三代测序中16srrna基因全长扩增的模板,剩余菌液加入等量50%的甘油于-80℃冰箱冻存。对43板96孔板的分离株进行编号。3.2分离株的16srrna基因全长扩增及测序参照armanhi等人的文章(armanhijsl,desouzars,dearaujolm,etal.multiplexampliconsequencingformicrobeidentificationincommunity-basedculturecollections.scientificreports,2016,6(1):29543),引用文中所设计的带有板标记(plate-barcode)(seqidnos.3-45中的第35至42位为板标记,引物名称依次为p1至p43)、行标记(r-barcode)(seqidnos.46-53中的第30至37位为行标记,引物名称依次为fr1至fr8)和列标记(c-barcode)(seqidnos.54-65中的第25至32位为列标记,引物名称依次为rc1至rc12)的barcode特异引物进行两轮pcr,两轮pcr的示意图见图1,其中,第一轮pcr上游引物如seqidno.66所示,其中第1至33位为正向转座酶序列,43板标记的3’端分别连接有部分反向转座酶序列(seqidno.67),以用于后续确定标记的位置。经过两轮pcr:第一轮以4128孔分离株的菌液分别为模板,使用带有板标记的下游引物,进行pcr扩增,经过1%琼脂糖凝胶电泳检测;第二轮pcr以第一轮pcr产物分别为模板,使用带有行标记的上游引物和带有列标记的下游引物,进行pcr扩增,经过1%琼脂糖凝胶电泳检测,得到大小为1500bp左右的pcr产物。对于第二轮pcr产物,从每孔中取5μl,将43板的第二轮pcr产物混合为一个样品,充分混合均匀后,共得到约21ml混合的pcr产物,由北京赛默百合公司在pacbiorsii平台进行16srrna基因全长序列三代测序分析。3.3分离株的16srrna基因全长序列三代测序数据分析根据pacbio’ssmrtportalv2.1.1中的rs_readsofinsert协议以及“minfullpasses2”和“minpredictedaccuracy90”的参数,通过环状共有序列(ccs)组装由pacbiorsii三代测序产生的4128孔分离株的原始序列数据。选取usableccss>10coverage的序列。根据标记(barcode)对ccs进行分析,利用usearch23软件包中的ublast算法鉴定正向转座酶、反向转座酶和1492r引物序列(seqidno.3),根据正向转座酶、反向转座酶和1492r引物序列三者各自的位置,确定标记的位置。基于确定了位置标记,利用usearch23软件包中的“search_oligodb”程序确定ccss的标记,从而对4128孔分离株的数据加以区分,并去掉标记信息:在标记分析过程中,通过严苛的选择条件保证序列位置的准确性:最大接受3个碱基的错配,不符合该标准的ccs将会被丢弃,最终得到与4128孔分离株对应的组别16srrna全长测序数据,其中由于某些孔的序列信息无法测出,因此这些孔内的分离株没有对应的数据,进而,在16srrna全长测序数据中少于4128组。利用usearch23软件包中的“usearch_global”程序和greengenes数据库,以相似性>97%为阈值对4128组16srrna全长测序数据进行过滤,得到4128组ccss。利用uchime程序中高质量无嵌合体参考数据库(www.drive5.com/uchime/rdp_gold.fa)针对所得到的各组有效数据去除嵌合体,在去除嵌合体的过程中,包括去除长度大于1600bp的ccs,如此得到4128组无嵌合体序列的有效数据。利用uparse软件(uparsev7.0.1001)对4128组有效数据分别进行otu聚类,按一致性97%以上的水平将有效数据聚类成为otus。使用rdpclassifier(version2.2)方法与greengene数据库对otus进行物种注释。经过以上处理之后共得到区分为分离株对应组别的33389条16srrna全长序列。将16srrna全长otus数据库与15个差异性关键otus数据进行本地blast,结果见表1。从表1可知,分析出5个一致性在96%以上的关键otus有5个,分别为otu137、otu4657、otu53、otu30和otu471其中,与otu137一致性为0.960494,与otu4657一致性为0.980488,otu53一致性为0.967901,otu30一致性为1,和otu471一致性为0.995349。根据表1中,基于这5个otus中的板标记、行标记和列标记的信息找出其所对应的96孔板编号(亦同时为带有板标记的引物名称)、行编号(亦同时为带有行标记的第二轮pcr上游引物名称)和列编号(亦同时为带有列标记的第二轮pcr下游引物名称)所对应的孔,进而找出重新编号为39、41、42、44和45的5个孔的潜在活性分离株。选择编号为39、41、42、44和45的5个孔的分离株作为初筛候选混合菌株。表1实施例4将编号为39、41、42、44和45分离株分别转接到5ml液体培养基(tsb培养基)的试管中于30℃培养12h,进行活化,然后转接到相应固体培养基(15cm)上,30℃培养12h,收集菌体,将收集的菌体重悬于20ml无菌水中。通过菌落计数的方法确定菌液浓度。以超纯水为阴性对照,测定分离株对暗黑鳃金龟具有杀虫活性。将胡萝卜擦丝,清水冲洗,晾干。取适量萝卜丝浸泡于菌液中,约20min后取出萝卜丝,均匀放于6孔生测板中,每孔约4-5条,剩余菌液拌于240g土中,得到的浓度为1×108cfu/g土。将拌好的土均匀分装至4个6孔生测板中,每孔接入1头初孵2日龄幼虫,每个重复接种24头,放至于温度25±1℃,rh(65±5)%,光照周期16l﹕8d的培养箱中饲养,每个浓度设置3个重复。其中,以无菌水(0cfu/g土)为阴性对照。每天检查光照、湿度、温度以及饲料是否霉变,是否有水蒸气的凝结。7d后调查死/活虫数,其中,阴性对照单个重复的平均活虫数为19,计算平均死亡率、根据阴性对照计算校正死亡率,结果见表2。表2分离株编号校正死亡率39—4155%4265%4451.6%45—接下来将编号为41、42和44的分离株进行划线纯化培养,挑取单克隆,用引物27f(seqidno.68)和1492r(seqidno.3)进行鉴定,分别获得与otu137、otu53、otu471相似性大于97%的单克隆,编号为41-6、42-3和44-7,作为候选菌株,在浓度为1×108cfu/g土和1×109cfu/g下进行生测,其他生测步骤同上。结果见表3。表3基于该方法,从4128孔分离株中,筛选出5孔分离株对暗黑鳃金龟幼虫进行活性初筛,结果3孔分离株均对暗黑鳃金龟幼虫有较好的杀虫活性,说明该方法不但可靠,而且大大降低了活性筛选的工作量,提高了筛选效率。序列表<110>中国农业科学院植物保护研究所<120>一种从环境中分离有益微生物的方法<130>lha2060580<160>68<170>siposequencelisting1.0<210>1<211>17<212>dna<213>人工序列(artificialsequence)<400>1cctaygggrbgcascag17<210>2<211>20<212>dna<213>人工序列(artificialsequence)<400>2ggactacnngggtatctaat20<210>3<211>59<212>dna<213>人工序列(artificialsequence)<400>3gtctcgtgggctcggagatgtgtataagagacagttaccgacgtaccttgttacgactt59<210>4<211>59<212>dna<213>人工序列(artificialsequence)<400>4gtctcgtgggctcggagatgtgtataagagacagattggacactaccttgttacgactt59<210>5<211>59<212>dna<213>人工序列(artificialsequence)<400>5gtctcgtgggctcggagatgtgtataagagacagtcgcatggataccttgttacgactt59<210>6<211>59<212>dna<213>人工序列(artificialsequence)<400>6gtctcgtgggctcggagatgtgtataagagacagagcgaaccttaccttgttacgactt59<210>7<211>59<212>dna<213>人工序列(artificialsequence)<400>7gtctcgtgggctcggagatgtgtataagagacagagcttcgactaccttgttacgactt59<210>8<211>59<212>dna<213>人工序列(artificialsequence)<400>8gtctcgtgggctcggagatgtgtataagagacaggtcagccgttaccttgttacgactt59<210>9<211>59<212>dna<213>人工序列(artificialsequence)<400>9gtctcgtgggctcggagatgtgtataagagacagtccagatagtaccttgttacgactt59<210>10<211>59<212>dna<213>人工序列(artificialsequence)<400>10gtctcgtgggctcggagatgtgtataagagacaggagagtccataccttgttacgactt59<210>11<211>59<212>dna<213>人工序列(artificialsequence)<400>11gtctcgtgggctcggagatgtgtataagagacaggctcacaattaccttgttacgactt59<210>12<211>59<212>dna<213>人工序列(artificialsequence)<400>12gtctcgtgggctcggagatgtgtataagagacagttgacgacataccttgttacgactt59<210>13<211>59<212>dna<213>人工序列(artificialsequence)<400>13gtctcgtgggctcggagatgtgtataagagacagcttagaacgtaccttgttacgactt59<210>14<211>59<212>dna<213>人工序列(artificialsequence)<400>14gtctcgtgggctcggagatgtgtataagagacagcggttcacataccttgttacgactt59<210>15<211>59<212>dna<213>人工序列(artificialsequence)<400>15gtctcgtgggctcggagatgtgtataagagacagcgataggcctaccttgttacgactt59<210>16<211>59<212>dna<213>人工序列(artificialsequence)<400>16gtctcgtgggctcggagatgtgtataagagacaggctatatcctaccttgttacgactt59<210>17<211>59<212>dna<213>人工序列(artificialsequence)<400>17gtctcgtgggctcggagatgtgtataagagacaggtcttcagctaccttgttacgactt59<210>18<211>59<212>dna<213>人工序列(artificialsequence)<400>18gtctcgtgggctcggagatgtgtataagagacagtagacaccgtaccttgttacgactt59<210>19<211>59<212>dna<213>人工序列(artificialsequence)<400>19gtctcgtgggctcggagatgtgtataagagacagtcagctgactaccttgttacgactt59<210>20<211>59<212>dna<213>人工序列(artificialsequence)<400>20gtctcgtgggctcggagatgtgtataagagacagtaagtcggctaccttgttacgactt59<210>21<211>59<212>dna<213>人工序列(artificialsequence)<400>21gtctcgtgggctcggagatgtgtataagagacaggctccttagtaccttgttacgactt59<210>22<211>59<212>dna<213>人工序列(artificialsequence)<400>22gtctcgtgggctcggagatgtgtataagagacagatggcctgataccttgttacgactt59<210>23<211>59<212>dna<213>人工序列(artificialsequence)<400>23gtctcgtgggctcggagatgtgtataagagacagttgcaagtataccttgttacgactt59<210>24<211>59<212>dna<213>人工序列(artificialsequence)<400>24gtctcgtgggctcggagatgtgtataagagacagcctagtaagtaccttgttacgactt59<210>25<211>59<212>dna<213>人工序列(artificialsequence)<400>25gtctcgtgggctcggagatgtgtataagagacagctaggatcataccttgttacgactt59<210>26<211>59<212>dna<213>人工序列(artificialsequence)<400>26gtctcgtgggctcggagatgtgtataagagacagtatgaacgttaccttgttacgactt59<210>27<211>59<212>dna<213>人工序列(artificialsequence)<400>27gtctcgtgggctcggagatgtgtataagagacagcttgtgcgataccttgttacgactt59<210>28<211>59<212>dna<213>人工序列(artificialsequence)<400>28gtctcgtgggctcggagatgtgtataagagacagcacgatggttaccttgttacgactt59<210>29<211>59<212>dna<213>人工序列(artificialsequence)<400>29gtctcgtgggctcggagatgtgtataagagacagacgtgcctttaccttgttacgactt59<210>30<211>59<212>dna<213>人工序列(artificialsequence)<400>30gtctcgtgggctcggagatgtgtataagagacagtgaactagctaccttgttacgactt59<210>31<211>59<212>dna<213>人工序列(artificialsequence)<400>31gtctcgtgggctcggagatgtgtataagagacagtattcagcgtaccttgttacgactt59<210>32<211>59<212>dna<213>人工序列(artificialsequence)<400>32gtctcgtgggctcggagatgtgtataagagacagtaatcggtgtaccttgttacgactt59<210>33<211>59<212>dna<213>人工序列(artificialsequence)<400>33gtctcgtgggctcggagatgtgtataagagacaggcgtccatgtaccttgttacgactt59<210>34<211>59<212>dna<213>人工序列(artificialsequence)<400>34gtctcgtgggctcggagatgtgtataagagacagcgtaagatgtaccttgttacgactt59<210>35<211>59<212>dna<213>人工序列(artificialsequence)<400>35gtctcgtgggctcggagatgtgtataagagacagctgttacagtaccttgttacgactt59<210>36<211>59<212>dna<213>人工序列(artificialsequence)<400>36gtctcgtgggctcggagatgtgtataagagacagacgatcatctaccttgttacgactt59<210>37<211>59<212>dna<213>人工序列(artificialsequence)<400>37gtctcgtgggctcggagatgtgtataagagacaggtaacggcttaccttgttacgactt59<210>38<211>59<212>dna<213>人工序列(artificialsequence)<400>38gtctcgtgggctcggagatgtgtataagagacagccatgcttataccttgttacgactt59<210>39<211>59<212>dna<213>人工序列(artificialsequence)<400>39gtctcgtgggctcggagatgtgtataagagacaggtacgcacataccttgttacgactt59<210>40<211>59<212>dna<213>人工序列(artificialsequence)<400>40gtctcgtgggctcggagatgtgtataagagacagttagagccataccttgttacgactt59<210>41<211>59<212>dna<213>人工序列(artificialsequence)<400>41gtctcgtgggctcggagatgtgtataagagacagataaggtcgtaccttgttacgactt59<210>42<211>59<212>dna<213>人工序列(artificialsequence)<400>42gtctcgtgggctcggagatgtgtataagagacagagtggcacttaccttgttacgactt59<210>43<211>59<212>dna<213>人工序列(artificialsequence)<400>43gtctcgtgggctcggagatgtgtataagagacagccagaagtgtaccttgttacgactt59<210>44<211>59<212>dna<213>人工序列(artificialsequence)<400>44gtctcgtgggctcggagatgtgtataagagacagctactagcgtaccttgttacgactt59<210>45<211>59<212>dna<213>人工序列(artificialsequence)<400>45gtctcgtgggctcggagatgtgtataagagacagtagcgttcctaccttgttacgactt59<210>46<211>51<212>dna<213>人工序列(artificialsequence)<400>46aatgatacggcgaccaccgagatctacactagatcgctcgtcggcagcgtc51<210>47<211>51<212>dna<213>人工序列(artificialsequence)<400>47aatgatacggcgaccaccgagatctacacctctctattcgtcggcagcgtc51<210>48<211>51<212>dna<213>人工序列(artificialsequence)<400>48aatgatacggcgaccaccgagatctacactatcctcttcgtcggcagcgtc51<210>49<211>51<212>dna<213>人工序列(artificialsequence)<400>49aatgatacggcgaccaccgagatctacacagagtagatcgtcggcagcgtc51<210>50<211>51<212>dna<213>人工序列(artificialsequence)<400>50aatgatacggcgaccaccgagatctacacgtaaggagtcgtcggcagcgtc51<210>51<211>51<212>dna<213>人工序列(artificialsequence)<400>51aatgatacggcgaccaccgagatctacacactgcatatcgtcggcagcgtc51<210>52<211>51<212>dna<213>人工序列(artificialsequence)<400>52aatgatacggcgaccaccgagatctacacaaggagtatcgtcggcagcgtc51<210>53<211>51<212>dna<213>人工序列(artificialsequence)<400>53aatgatacggcgaccaccgagatctacacctaagccttcgtcggcagcgtc51<210>54<211>47<212>dna<213>人工序列(artificialsequence)<400>54caagcagaagacggcatacgagattcgccttagtctcgtgggctcgg47<210>55<211>47<212>dna<213>人工序列(artificialsequence)<400>55caagcagaagacggcatacgagatctagtacggtctcgtgggctcgg47<210>56<211>47<212>dna<213>人工序列(artificialsequence)<400>56caagcagaagacggcatacgagatttctgcctgtctcgtgggctcgg47<210>57<211>47<212>dna<213>人工序列(artificialsequence)<400>57caagcagaagacggcatacgagatgctcaggagtctcgtgggctcgg47<210>58<211>47<212>dna<213>人工序列(artificialsequence)<400>58caagcagaagacggcatacgagataggagtccgtctcgtgggctcgg47<210>59<211>47<212>dna<213>人工序列(artificialsequence)<400>59caagcagaagacggcatacgagatcatgcctagtctcgtgggctcgg47<210>60<211>47<212>dna<213>人工序列(artificialsequence)<400>60caagcagaagacggcatacgagatgtagagaggtctcgtgggctcgg47<210>61<211>47<212>dna<213>人工序列(artificialsequence)<400>61caagcagaagacggcatacgagatcctctctggtctcgtgggctcgg47<210>62<211>47<212>dna<213>人工序列(artificialsequence)<400>62caagcagaagacggcatacgagatagcgtagcgtctcgtgggctcgg47<210>63<211>47<212>dna<213>人工序列(artificialsequence)<400>63caagcagaagacggcatacgagatcagcctcggtctcgtgggctcgg47<210>64<211>47<212>dna<213>人工序列(artificialsequence)<400>64caagcagaagacggcatacgagattgcctcttgtctcgtgggctcgg47<210>65<211>47<212>dna<213>人工序列(artificialsequence)<400>65caagcagaagacggcatacgagattcctctacgtctcgtgggctcgg47<210>66<211>49<212>dna<213>人工序列(artificialsequence)<400>66tcgtcggcagcgtcagatgtgtataagagacagagagtttgatcmtggc49<210>67<211>30<212>dna<213>人工序列(artificialsequence)<400>67gtctcgtgggctcggagatgtgtataagag30<210>68<211>20<212>dna<213>人工序列(artificialsequence)<400>68agagtttgatcmtggctcag20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1