一种抗草甘膦转基因大豆的培育方法与流程
一种抗草甘膦转基因大豆的培育方法
【技术领域】
[0001]
本发明涉及植物基因工程技术领域,具体涉及一种抗草甘膦转基因大豆的培育方法。
背景技术:
[0002]
大豆(glycine max(l.).merr)起源于中国,是世界上重要的油料作物和经济作物,也是食用植物油与植物蛋白质的主要来源。杂草作为农业生态系统中的重要组成部分,可以与作物竞争光、水、养分等资源,从而使作物减产。因此,大豆田间杂草的有效防除是大豆稳产高产的关键因素之一。传统的人工除草包括手工拔草和使用简单农具除草等,耗时耗力,功效低且不能大面积及时防除。化学除草剂的使用大大提高了农田杂草的防治效率,但是传统除草剂如氯嘧磺隆、甲磺隆等在土壤中残留严重、污染环境,限制了这些除草剂的使用范围。
[0003]
草甘膦(glyphosate)为内吸传导型、广谱灭生性除草剂,具有杀草谱广、高效、低毒、无残留等优点,尤其对人畜毒害小,是全球应用范围最广的除草剂之一。但作为一种非选择性除草剂,草甘膦在杀灭杂草的同时也伤害大豆,因此通过转基因技术培育抗草甘膦转基因作物,赋予作物抗草甘膦特性,是保护作物不受草甘膦伤害,提高杂草综合防治效率的重要途径之一。已有的研究和转基因作物商业化发展历史证明大豆对草甘膦除草剂的耐受性将是对于杂草防除、管理的有用性状。
[0004]
为赋予农作物草甘膦的耐受性,研究者集中于向植物体内导入能增加草甘膦耐受性的基因,如epsps,epsps基因通常来自于微生物,其编码酶的结构特性使得草甘膦不能与其结合,因而在草甘膦存在的情况下仍然能够保持了他们的催化活性(pct/cn03/00651)。当前全球商业化种植的草甘膦抗性转基因作物绝大多数为针对epsps所设计。
[0005]
在植物组织中n-乙酰转移酶(gat)能够通过n-乙酰化作用使得草甘膦被有效的降解,从而失去除草剂活性,且乙酰化的草甘膦不能与植物体内的epsps有效的结合,从而赋予植物对草甘膦的耐受性(zl 200510086626.x)。此外,由于草甘膦的降解,利用n-乙酰化的手段培育转基因作物可以在植物整个生长周期都可以施用草甘膦,不受生长发育阶段的限制。
[0006]
当前,关于抗草甘膦基因导入大豆的研究比较广泛,但不同位点的插入会影响大豆其他基因的功能,因此有必要结合抗草甘膦基因的合成,实现不同位点的插入,满足外源基因在其后代可以稳定遗传且具有草甘膦除草剂耐受性,以减少生产管理中的劳动力成本,提高种植效益。
技术实现要素:
[0007]
基于现有技术的不足,本发明提供一种抗草甘膦转基因大豆的培养方法,通过外源基因载体的优化,并插入到不同位点,获得满足外源基因在其后代可以稳定遗传且具有草甘膦除草剂耐受性的转基因大豆。
[0008]
鉴于此,本发明的目的在于,提出一种抗草甘膦转基因大豆的培育方法,将外源基因dna片段插入天隆1号大豆基因组第19号染色体内得到转基因大豆,插入位点的左边界侧翼序列如seq id no:1所示,插入位点的右边界侧翼序列如seq id no:2所示;所述外源基因dna片段包含草甘膦抗性基因。
[0009]
进一步地,所述外源基因dna片段核苷酸序列如seq id no:3所示。
[0010]
进一步地,所述外源基因dna片段用抗草甘膦基因i.variabilis-mepsps构建植物表达载体所得。
[0011]
进一步地,所述外源dna片段插入过程为通过农杆菌介导的遗传转化方法,将外源dna片段导入到目的大豆受体中。
[0012]
本发明的另一个目的在于,提供上述培育方法获得的转基因大豆外源基因的检测方法,包括利用gmcyp2基因作为内参基因,设计引物gmcyp2-rtf与gmcyp2-rtr进行质量检测,设计引物iva-epsps-rtf与iva-epsps-rtr进行外源基因表达量检测;所述引物gmcyp2-rtf与gmcyp2-rtr的核苷酸序列如seq id no:4-5所示;所述设计引物iva-epsps-rtf与iva-epsps-rtr的核苷酸序列如seq id no:6-7所示。
[0013]
本发明的还有一个目的在于,提供上述培育方法获得的转基因大豆外源基因位点的分析方法,利用反向pcr方法进行两轮pcr分析,然后进行位点验证得到侧翼序列,第一轮pcr的引物核苷酸序列如seq id no:8-9所示;第二轮pcr的引物如seq id no:10-11所示。
[0014]
根据本发明提供的一个实施例,所述位点验证利用引物gm1-v1f与gm17-glbr进行左边界侧翼序列验证,利用gm1-v2f与gm17-grbr进行右边界侧翼序列验证;所述引物gm1-v1f与gm17-glbr的核苷酸序列如seq id no:12-13所示;所述引物gm1-v2f与gm17-grbr的核苷酸序列如seq id no:14-15所示。
[0015]
根据本发明提供的一个实施例,所述左边界侧翼序列验证结果在520bp处有特异性条带,右边界侧翼序列验证结果在592bp处有特异性条带。
[0016]
相比现有技术,本发明的有益效果为:
[0017]
1)本发明将人工合成的抗草甘膦基因i.variabilis-epsps*导入大豆受体品种天隆1号的基因组中,外源基因在其后代可以稳定遗传且具有草甘膦除草剂耐受性,可减少生产管理中的劳动力成本,提高种植效益。
[0018]
2)本发明抗草甘膦基因i.variabilis-epsps*在大豆基因组中的位置不同于任何其他任何含有类似基因的抗草甘膦大豆,并且外源基因在大豆基因组中的插入位点不影响大豆其他基因的功能的发挥。
[0019]
3)本发明培育的抗草甘膦转基因大豆材料可以通过杂交、回交等常规育种方式,将抗草甘膦基因导入给其它大豆品种,进一步培育抗草甘膦除草剂转基因新品系或新品种。
【附图说明】
[0020]
为了更清楚地说明本发明实施例的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,应当理解,以下附图仅示出了本发明的某些实施例,因此不应被看作是对范围的限定,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
[0021]
图1是pcambia1300载体图图谱。
[0022]
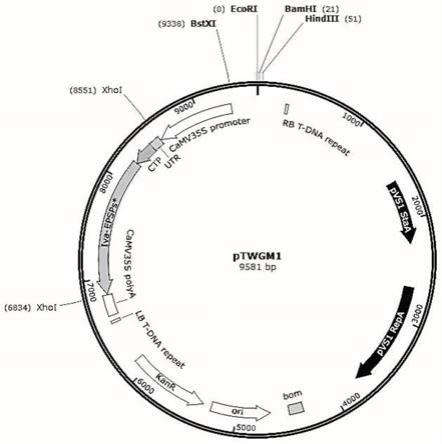
图2是本发明植物表达载体ptwgm1的图谱。
[0023]
图3是ptwgm1-7中iva-epsps*的rt-pcr检测结果。
[0024]
图4是转基因纯合株系对草甘膦的抗性表现对比结果。
[0025]
图5为ptwgm1-7左边界与右边界侧翼序列验证结果。
[0026]
图6为ptwgm1-7左边界侧翼序列ncbi序列比对结果。
[0027]
图7为ptwgm1-7右边界侧翼序列ncbi序列比对结果。
【具体实施方式】
[0028]
以下实例用于说明本发明,但不限制本发明的范围。在不背离本发明精神和实质的前提下,对本发明的方法、步骤或条件所作的修改或替换,均属于本发明的范围。
[0029]
本发明涉及的抗草甘膦转基因大豆的培育方法,将外源dna片段插入目的大豆基因组,通过单拷贝植株纯合株系筛选,得到草甘膦抗性植株ptwgm1-7。通过外源基因的检测及插入位点左右侧翼序列分析验证,其基因组外源基因的插入部位为第19号染色体,且替换掉了55bp的碱基序列,通过后代种植后,表现出优秀的草甘膦的抗性,并且正常生长。
[0030]
根据常用的技术手段,本发明培育的抗草甘膦转基因大豆材料可以通过杂交、回交等常规育种方式,将抗草甘膦基因导入给其它大豆品种,进一步培育抗草甘膦除草剂转基因新品系或新品种。
[0031]
本发明所使用的外源基因为i.variabilis-epsps*,由华中农业大学刘子铎和林拥军教授合成并取得(专利申请号:201610317483.7;林拥军,崔莹,刘子铎:一种修饰的抗草甘膦基因及抗草甘膦水稻的培育方法:公开号为cn107129993a)。i.variabilis-epsps*基因是通过序列比对从新发现的放射线菌(isoptericola variabilis)中发现有一段序列中编码的蛋白与i型epspss相似,在大肠杆菌(e.coli)中表达发现对草甘膦具有极高的抗性,其酶学特性与i型epspss相似,这可能是一种新型的抗草甘膦类型。i.variabilis-epsps*基因序列与i型的epspss代表基因来源于e.coli相似性为35%,与ii型epspss典型的根癌农杆菌(a.tumefaciens)cp4和金黄色葡萄球菌(s.aureus)序列相似性分别为29%和26%,通过两类epsps基因氨基酸多序列比对和进化分析,申请人将i.variabilis-epsps*归类为i型的epsps基因,但属于一个新分支。i.variabilis-epsps*基因的原始长度为1374bp,编码458个氨基酸。通过利用密码子优化软件,并根据密码子偏好性进行了优化。其完整的基因序列见相关的发明专利。
[0032]
实施例1.植物表达载体的构建
[0033]
本发明的转化载体的构建过程如下:利用xhoi酶切去除pcambia1300载体中的hpt基因,然后把两端携带xhoi粘性末端的utr-ctp-epsps片段与去除了hpt基因的载体进行连接,获得最终的表达载体ptwgm1(图谱如图2所示)。epsps基因全长1374bp,在其5’端添加了100bp的utr序列与228bp的叶绿体定位信号肽ctp。最终的植物表达载体ptwgm1的质粒,核苷酸序列见seq id no:1,通过电转化方式导入到大肠杆菌dh5α中,经测序验证后,再导入到农杆菌菌株eha105中形成转化的工程菌株,在-80℃冰箱冷冻保存备用。
[0034]
实施例2.农杆菌介导的大豆遗传转化
[0035]
2.1大豆种子氯气消毒
[0036]
挑选饱满、无病斑且种皮完好的大豆种子,在通风橱里用盐酸和次氯酸钠反应产生的氯气灭菌3h,然后放入无菌操作台通风30min,然后用无菌水充分浸泡过夜。
[0037]
2.2种子浸泡过夜
[0038]
选取消毒后的大豆种子,无菌水充分浸泡过夜。
[0039]
2.3分离外植体
[0040]
将浸泡过夜的大豆种子沿着胚生长的方向横切一分为二、弃种皮、切除部分顶芽并顺着子叶轴方向在靠近子叶节处垂直划出一条创口,由此每个无菌大豆种子可得到两个用于遗传转化的子叶节外植体。
[0041]
2.4农杆菌的培养
[0042]
在50ml含有50mg/l卡那霉素的lb培养基(10g/l蛋白胨,5g/l酵母粉,10g/l氯化钠,ph 7.0)中,接入50μl保存的农杆菌,在摇床(28℃,200rpm)中培养2d,活化农杆菌。
[0043]
2.5农杆菌浸染
[0044]
将活化好的农杆菌菌液,离心收集菌体,然后用ccm液体培养基重悬至od≈0.6,然后将制备好的外植体放入重悬后的浸染液中,28℃恒温摇床200rpm震荡30min。
[0045]
2.6共培养
[0046]
将浸染后的外植体近轴创口面朝下置于铺有无菌滤纸的ccm固体培养基,28℃,暗培养3d。
[0047]
2.7水洗除菌、转sim诱导培养
[0048]
将暗培养3d后的外植体用无菌水冲洗三次、sim液体培养基冲洗三次,然后切除每个外植体伸长顶芽,将得到的新的外植体插入含有一定浓度筛选剂的sim固体芽诱导培养基,28℃16h/8h光照培养2周。
[0049]
2.8抗性丛生芽继代培养
[0050]
将抗性丛生芽组织转移至新的含有一定浓度筛选剂的sim培养约2周,然后更换培养基重复继代1次。
[0051]
2.9抗性丛生芽转sem培养基伸长培养
[0052]
切除抗性丛生芽基部黑色组织,将休整过的抗性丛生芽组织转入含有一定浓度筛选剂的的sem固体培养基28℃16h/8h光照培养4周。
[0053]
2.10伸长芽继代培养
[0054]
切除抗性丛生芽团白色、黄色或黑色部分以及基部黑色组织,将休整过的丛生芽绿色组织转入新的含有一定浓度筛选剂的sem再次培养4周,然后更换培养基重复继代1次。
[0055]
2.11伸长芽转rm生根
[0056]
当大豆再生幼苗培养至3~4cm高时,将大豆幼苗从基部切除并转入rm培养基生根。
[0057]
2.12培养基配制方法
[0058]
1)共培养基ccm(1l):
[0059]
b5粉0.321g,mes 3.9g,100
×
b5有机9ml,琼脂7g,蔗糖30g,dtt 1ml、na2s2o31ml、6-ba 1.67ml、as 2ml、l-cys 4ml和ga3 100μl。
[0060]
2)芽诱导培养基sim(1l):
[0061]
b5粉3.21g,mes 0.59g,琼脂8g,蔗糖30g,特美汀1ml,6-ba 1120μl,头孢霉素400μ
l和一定浓度筛选剂(草甘膦)。
[0062]
3)芽伸长培养基sem(1l):
[0063]
ms粉4.43g,mes 0.59g,琼脂8g,蔗糖30g,glu 1ml,asp 1ml,zr 1ml,特美汀1ml,ga3 600μl,头孢霉素400μl,iaa 100μl和一定浓度筛选剂(草甘膦)。
[0064]
4)生根培养基rm(1l):
[0065]
ms粉2.215g,mes 0.59g,100
×
b5有机9ml,琼脂8g,蔗糖20g,特美汀1ml,glu 1ml,asp 1ml,iba 1ml和头孢霉素400μl。
[0066]
实施例3.转基因再生植株的pcr阳性检测
[0067]
采用ctab法提取大豆叶片总dna(文献:murry&thompson,1980)。pcr引物的设计利用primer premier 5软件完成。用于i.variabilis-epsps*基因(后续简写为iva-epsps)扩增的两条引物分别是:
[0068]
iva-epspsf:5
’-
attagcgctagggacgtgag-3’,与
[0069]
iva-epspsr:5
’-
atacgctcccacatcctgtc-3’。
[0070]
pcr产物大小是593bp。pcr反应体系:20ng模板dna,2μl 10
×
pcr buffer,0.3μl dntp(10mm),0.3μl iva-epspsf引物(10μm),0.3μl iva-epspsr引物(10μm),1u rtaq,补ddh2o至20μl。pcr反应程序:94℃预变性5min,94℃变性30s,57℃退火30s,72℃延伸30s;重复32个循环;72℃,5min;25℃,2min。扩增产物在0.8%的琼脂糖凝胶上进行电泳检测。对所有t0代再生植株进行pcr阳性检测,一共检测t0代转化植株40株,其中29株转化植株为iva-epsps*阳性植株。
[0071]
实施例4.转基因植株中外源基因的拷贝数检测
[0072]
利用southern blot技术对所有的pcr检测阳性植株进行拷贝数检测。大豆植株的叶片总dna采用ctab法提取(参见文献:murry&thompson,1980),southern blot具体的实验方法参见roche公司的地高辛标记方法应用手册。总dna用hindiii或者saci酶切,iva-epsps*基因的pcr片段作为杂交探针,地高辛标记以后进行杂交检测。根据southern blot结果,3个t0代转基因家系为单拷贝转基因家系。
[0073]
实施例5.单拷贝植株纯合株系筛选
[0074]
3个单拷贝大豆转基因家系在田间种植t1代,分单株收种,然后每个单株挑选60粒种子种植t2代,对t2代苗期植株喷施草甘膦。根据对草甘膦表现出抗性表现的后代植株比例判断其对应的t1代植株是否为纯合株系:纯合株系的后代100%对草甘膦表现出抗性,杂合株系的后代75%对草甘膦表现出抗性,阴性植株的所有后代对草甘膦表现出敏感(喷施草甘膦后全部死亡)。经过纯合株系筛选,ptwgm1-7单拷贝家系对草甘膦表现出较高的抗性水平。
[0075]
实施例6:纯合株系中外源基因的rt-pcr表达量检测
[0076]
采用全式金的transzol试剂提取大豆叶片中的总rna,具体实验步骤参考其说明书。rna反转录采用以下步骤:
[0077]
1)取2μg rna,加入1μl dnasei buffer(with mg
2+
,thermo),1μl dnasei,补水至10μl,37℃,30min;
[0078]
2)加入1μl 50mm edta,混匀,65℃变性10min;
[0079]
3)加入2μl dntps(10mm)和1μl oligo(dt)
15
(50μm),混匀,65℃变性5min,迅速冰
浴5min;
[0080]
4)稍作离心,加入5μl 5
×
first-strand buffer、2μl 0.1m dtt、1μl rnaseout
tm recombinant ribonuclease inhibitor(40u/μl)和1μl m-mlv反转录酶,2μl depc水,混匀,37℃孵育50min;
[0081]
5)70℃失活15min,终止反转录反应,获得的cdna加depc水至100μl备用。
[0082]
利用gmcyp2基因作为内参基因,设计引物gmcyp2-rtf与gmcyp2-rtr对cdna进行质量检测,设计引物iva-epsps-rtf与iva-epsps-rtr检测外源基因表达量。pcr反应体系:1μl cdna,2μl 10
×
pcr buffer,0.3μl dntp(10mm),0.3μl f引物(10μm),0.3μl r引物(10μm),1u rtaq,补ddh2o至20μl。pcr反应程序:94℃预变性5min,94℃变性30s,57℃退火30s,72℃延伸30s;重复30个循环;72℃,5min;25℃,2min。ptwgm1-7中iva-epsps*的rt-pcr检测结果如图3所示(n:天隆1号对照;1:ptwgm1-7纯合株系),外源基因epsps*在转化体ptwgm1-7中高水平表达。
[0083]
表1:rt-pcr检测引物
[0084][0085]
实施例7:转基因纯合株系在田间对草甘膦抗性检测
[0086]
在田间种植ptwgm1-7纯合株系后代,受体品种天隆1号作为对照。6周后,用生产剂量的4倍草甘膦浓度(3600g/ha,对应的喷施浓度3600mg/l)对转基因纯合株系及对照材料行进喷施处理,草甘膦处理2周后,转基因株系生长正常而对照材料天隆1号表现出敏感,如图4所示,ck为对照材料天隆1号。
[0087]
实施例8:ptwgm1-7插入位点分析
[0088]
利用反向pcr方法对ptwgm1-7的插入位点进行分析。取2μg基因组dna,用hindiii或者saci进行酶切,75℃失活10min,然后用t4 dna连接酶进行连接。对连接产物进行巢式pcr:取0.5μl连接产物进行第一轮pcr,反应体系为:0.5μl连接产物,10μl 2
×
kod buffer,3μl dntp(2mm),0.3μl 10μm gm1-vb1(序列见表2),0.3μl 10μm iva-1(序列见表2),0.4μl kod,补ddh2o至20μl。pcr反应程序:98℃预变性3min;98℃变性30s,55℃退火30s,68℃延伸3min,30个循环;68℃,5min;25℃,2min。第二轮pcr反应体系为0.5μl第一轮pcr产物,10μl 2
×
kod buffer,3μl dntp(2mm),0.3μl 10μm gm1-vb1(序列见表2),0.3μl 10μm iva-2(序列见表2),0.4μl kod polymerase,补ddh2o至20μl。pcr反应程序:98℃预变性3min;98℃变性30s,55℃退火30s,68℃延伸3min,35个循环;68℃,5min;25℃,2min。对第二轮pcr产物进行琼脂糖凝胶电泳检测,胶回收后送测序,测序结果与ncbi数据库进行比对分析,确定t-dna在大豆基因组中的插入位点。
[0089]
表2:插入位点分析引物
[0090][0091]
实施例9:ptwgm1-7中外源基因插入位点验证
[0092]
根据实施例7中的侧翼序列分离结果,设计引物(表3)进行特异性pcr验证分析。利用引物gm1-v1f与gm17-glbr对ptwgm1-7基因组dna样品进行pcr扩增,验证t-dna插入位点左边界的侧翼序列,利用引物gm1-v2f与gm17-grbr对ptwgm1-7基因组dna样品进行pcr扩增,验证t-dna插入位点右边界的侧翼序列。pcr反应体系:20ng模板dna,2μl 10
×
pcr buffer,0.3μl dntp(10mm),0.3μl gm1-v1f(或gm1-v2f)引物(10μm),0.3μl gm17-glbr(或gm17-grbr)引物(10μm),1u rtaq,补ddh2o至20μl。pcr反应程序:94℃预变性5min,94℃变性30s,57℃退火30s,72℃延伸30s;重复32个循环;72℃,5min;25℃,2min。ptwgm1-7扩增结果如图5所示(5a为左边界侧翼序列验证;5b为右边界侧翼序列验证,m:dl2000 dna marker;n:天隆1号对照;1为ptwgm1-7纯合株系)。
[0093]
左边界验证结果在520bp处有特异性条带,右边界验证结果在592bp处有特异性条带。t-dna两端的侧翼序列在ncbi网站与williams 82的基因组序列进行比对,结果显示,ptwgm1-7转化体中,t-dna插入到大豆的第19号染色体(图6与图7),且由于t-dna的插入,导致了大豆基因组中缺失了55bp。
[0094]
表3:插入位点验证引物
[0095][0096]
>ptwgm1-7左边界侧翼序列如下:
[0097]
actgaatagactcatccaggctgatttttaaggcattgatatgaacacagttagacaaatgaaatgttgatgtaagaaaaaaaatttgaattgtaatttgattttagtaaattttgtttatgagaagttttagtaacatattctttaatacaatttttattatgattaaaatttattaaaaaatacaaaaataattaatagagtctttaaatgtgagacttataaagttttgtatttttaaataatttttaatcaataataataaagtatgtttaaaagagttttaaatagcatattgttagcaattctattactatgtctatt
[0098]
>ptwgm1-7右边界侧翼序列如下:
[0099]
tctttgcaaaggcaaagacaccgaagttgagaaatattttcgttgtcttcataaaaaaaatcgttgtcattcaaaatttctataaagctctcggatggtcgaatgcagacttgttgtgcaatcacgtgtcttttgttcaatcacgt
gtcttttgttcatattaatggtgccgacataagtaagggtaaaaatagatcataccatccatcataaacaagttaaacttagattttttttaaaatatttttaataaaaaatgtgagactctagactttaaataaaaatcttttaagtttgataagttgacatgtttatataataatatataattatatattatttaattttaatatataattattaatattgataataagtttataataaaaataatgtgacg
[0100]
本发明并不仅仅限于说明书和实施方式中所描述,因此对于熟悉领域的人员而言可容易地实现另外的优点和改进,故在不背离权利要求及等同范围所限定的一般概念的精神和范围的情况下,本发明并不限于特定的细节、代表性的方案和这里示出与描述的图示示例。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1