一种具有启动、终止双功能调控元件的构建方法、双功能元件库和应用
本发明涉及合成生物学技术领域,尤其涉及一种具有启动、终止双功能调控元件的构建方法、双功能元件库和应用。
背景技术:
复杂的化合物经常被应用于溶剂、燃料、聚合物、纺织品、营养品、香料和药品。然而,由于工业化对环境的污染、温室气体影响造成的气候变化以及原油短缺的担忧,促使人们寻找新的、可持续的方式来供应合成化学品的供应。代谢工程与合成生物学的出现为研究和发展生物技术提供了崭新思路,代谢途径和遗传电路通常被引入微生物中,以产生化学物质或实现新的功能。酵母作为传统工业微生物能够以安全可靠的方式生产高值天然产物、化工产品和材料等。这类实验通常需要将异源基因电路或代谢途径在酵母体内表达,实现目标产物的合成。
基因电路的构建需要一个完整的基因表达盒-包括启动子、基因、终止子,而代谢途径的构建需要将不同表达盒连接到一起,虽然化学方法可以合成一定长度的dna片段,但为了更大表达模块的组装仍然需要dna组装技术,包括goldengate方法、in-fusion技术、gibson组装、dnaassembler等。现有技术中常使用启动子cyc1p、tys1p等,终止子adh1t、pdalt等,但单独的启动子和终止子用于基因电路或途径组装时常常耗时较长,且当组装途径较长时,组装效率也有限。
技术实现要素:
有鉴于此,本发明的目的在于提供一种具有启动、终止双功能调控元件的构建方法、双功能元件库和应用,采用本发明提供的构建方法构建得到的具有启动、终止双功能调控元件应用到途径构建中,可以简化途径构建过程,增强组装。
为了实现上述发明目的,本发明提供以下技术方案:
本发明提供了一种具有启动、终止双功能调控元件的构建方法,将启动序列和终止序列按照先终止后启动的原则进行组装;
所述启动序列包括上游活性序列和核心启动子序列,所述上游活性序列包括mig1/mig2结合位点、rap1结合位点、gcr1结合位点、weakmig1结合位点、gc框、caat框和八聚体框;所述核心启动子序列包括tata框、bre元件、mte元件和inr元件;
所述终止序列包括效率元件、位置元件和polya。
优选的,所述mig1/mig2结合位点的核苷酸序列如seqidno.1所示;
所述rap1结合位点的核苷酸序列如seqidno.2所示;
所述gcr1结合位点的核苷酸序列如seqidno.3所示;
所述weakmig1结合位点的核苷酸序列如seqidno.4所示;
所述gc框的核苷酸序列为gggcgg;
所述caat框的核苷酸序列为ccaatct;
所述八聚体框的核苷酸序列为atgcaaat。
优选的,所述tata框的核苷酸序列为tatataaa;
所述bre元件的核苷酸序列为ggacgcc;
所述mte元件的核苷酸序列如seqidno.5所示;
所述inr元件的核苷酸序列为ttaatat。
优选的,所述效率元件的核苷酸序列为tatatata,所述位置元件的核苷酸序列为aataaa。
优选的,将所述核心启动子序列连接在上游活性序列前。
优选的,将所述终止序列连接在核心启动子序列前。
优选的,将所述终止序列插入在核心启动子序列和上游活性序列之间。
本发明还提供了一种基于上述技术方案所述的构建方法构建得到的具有启动、终止双功能调控元件,所述具有启动、终止双功能调控元件的核苷酸序列如seqidno.6所示。
本发明还提供了一种基于上述技术方案所述的构建方法得到的双功能元件库,将启动序列和终止序列按照先终止后启动的原则进行组装,调节所述启动序列和终止序列中各种序列的排列顺序和间隔区,得到双功能元件库。
本发明还提供了上述技术方案所述的具有启动、终止双功能调控元件或上述技术方案所述的双功能元件库在番茄红素途径构建中的应用。
本发明提供了一种具有启动、终止双功能调控元件的构建方法、双功能元件库和应用,将启动序列和终止序列按照先终止后启动的原则进行组装;所述启动序列包括上游活性序列和核心启动子序列,所述上游活性序列包括mig1/mig2结合位点、rap1结合位点、gcr1结合位点、weakmig1结合位点、gc框、caat框和八聚体框;所述核心启动子序列包括tata框、bre元件、mte元件和inr元件;所述终止序列包括效率元件、位置元件和polya。采用本发明提供的构建方法构建得到的具有启动、终止双功能调控元件应用到途径构建中,由于构建过程中减少了片段数量,简化代谢途径构建过程,组装效率相比于现有技术提高了约18%。
附图说明
图1为核心启动子的功能验证结果;
图2为启动子的功能验证结果;
图3为终止子的功能验证结果;
图4为双功能元件的功能验证结果;
图5为不同强度的双功能元件库。
具体实施方式
本发明提供了一种具有启动、终止双功能调控元件的构建方法,将启动序列和终止序列按照先终止后启动的原则进行组装;所述启动序列包括上游活性序列和核心启动子序列,所述上游活性序列包括mig1/mig2结合位点、rap1结合位点、gcr1结合位点、weakmig1结合位点、gc框、caat框和八聚体框;所述核心启动子序列包括tata框、bre元件、mte元件和inr元件;所述终止序列包括效率元件、位置元件和polya。
在本发明中,所述mig1/mig2结合位点的核苷酸序列如seqidno.1所示;所述rap1结合位点的核苷酸序列如seqidno.2所示;所述gcr1结合位点的核苷酸序列如seqidno.3所示;所述weakmig1结合位点的核苷酸序列如seqidno.4所示;所述gc框的核苷酸序列为gggcgg;所述caat框的核苷酸序列为ccaatct;所述八聚体框的核苷酸序列为atgcaaat。
seqidno.1:gatcgtagccccggatttac。
seqidno.2:gatcgtacacctggacatac。
seqidno.3:gatcgtagagcttccactac。
seqidno.4:gatcgtagccccacaaatac。
在本发明中,所述tata框的核苷酸序列为tatataaa;所述bre元件的核苷酸序列为ggacgcc;所述mte元件的核苷酸序列如seqidno.5所示;所述inr元件的核苷酸序列为ttaatat。
seqidno.5:cgagccgagca。
在本发明中,所述效率元件的核苷酸序列为tatatata,所述位置元件的核苷酸序列为aataaa。
本发明优选将所述核心启动子序列连接在上游活性序列前。本发明优选将所述终止序列连接在核心启动子序列前,或者将所述终止序列插入在核心启动子序列和上游活性序列之间。上述连接或插入后,转录水平使其启动和终止功能完整且互不影响。
本发明还提供了一种基于上述技术方案所述的构建方法构建得到的具有启动、终止双功能调控元件,所述具有启动、终止双功能调控元件的核苷酸序列如seqidno.6所示,具体如下:
seqidno.6:
aagacagtttatatataacgatagtaataaattattcagttgataatctcgtttcaaagcagacgtgcctaagtgataatcgatcgtagccccggatttacgtaagttctggatcgtacacctggacatacagattgtgatggcaatctcgattacactgatcgtagagcttccactactaaatcggtgatcgtagccccacaaataccgtgatactgtattgtattttcacaggacgcctatataaaactcttgttttcttcttttctcttaatattcttttcgagccgagcactacggaccg。
本发明还提供了一种基于上述技术方案所述的构建方法得到的双功能元件库,将启动序列和终止序列按照先终止后启动的原则进行组装,调节所述启动序列和终止序列中各种序列的排列顺序和间隔区,得到双功能元件库。
本发明还提供了上述技术方案所述的具有启动、终止双功能调控元件或上述技术方案所述的双功能元件库在番茄红素途径构建中的应用。
为了进一步说明本发明,下面结合实例对本发明进行详细地描述,但不能将它们理解为对本发明保护范围的限定。
实施例1
启动功能的设计与验证。
通过ncbi数据库分析大量酿酒酵母启动子序列信息得知:酵母组成型启动子由上游活性序列和核心启动子序列组成,其中上游活性序列包含大量转录因子结合位点,选用酵母转录因子结合位点mig1/mig2结合位点序列为:gatcgtagccccggatttac、rap1结合位点序列为:gatcgtacacctggacatac、gcr1结合位点序列为:gatcgtagagcttccactac、weakmig1结合位点序列为:gatcgtagccccacaaatac,转录因子结合位点可有效提高启动子转录水平。及优选辅助rnaii聚合酶与dna结合的转录因子结合位点:gc框:gggcgg、caat框:ccaatct、八聚体框:atgcaaat;核心启动子序列包含:tata框tatataaa位于起始位点上游-25~-30bp处、bre元件ggacgcc位于起始位点上游-37~-32bp处、mte元件cgagccgagca位于起始位点下游+11~+18bp处和inr元件:ttaatat组成。
本实施例所设计的启动序列包含上游活性序列uas及核心启动子序列;其中连接序列的变化、转录因子结合位点的选择不同及排列顺序不同均会造成启动子强度发生变化。本实施例仅取一例作为讲解对象。
上游活性序列uas(seqidno.7):agtgataatcgatcgtagccccggatttacgtaagttctggatcgtacacctggacatacagattgtgatggcaatctcgattacactgatcgtagagcttccactactaaatcggtgatcgtagccccacaaataccgtgatactg,下划线序列依次为上述酵母转录因子结合位点mig1/mig2、rap1、gcr1、weakmig1。其余序列为随机连接序列,以确保上游活性序列与酵母同源性最低,以防止该序列在酵母体内发生同源重组,从而不能正常行使其功能。
核心启动子序列(seqidno.8):
tattgtattttcacaggacgcctatataaaactcttgttttcttcttttctctta+1atattcttttcgagccgagcactacggaccg,下划线序列依次为bre元件、tata框、inr元件(+1处为转录起始位点)及mte元件。其余序列为t-rich随机连接序列,选用t-rich序列可使rnaⅱ聚合酶更好的与dna序列结合。
将上述上游活性序列与核心启动子序列直接连接形成人工启动子序列,化学合成后扩增获得启动子序列片段(以引物1、引物2扩增获得)。
引物序列为:
引物1(seqidno.9):cgggatccagtgataatcgatcg,下划线序列为bamhi酶切位点的重叠序列。
引物2(seqidno.10):
ttttccttttgcggccgccggtccgtagtgctcggc,下划线序列为noti酶切位点的重叠序列。
验证载体质粒1的构建:
从genebank基因库中查询获得报告基因egfp片段,化学合成后扩增获得egfp基因片段(以引物3、引物4扩增获得)。
引物序列为:
引物3(seqidno.11):
ataagaatgcggccgcatggtgagcaagggagaagaac,下划线序列为noti酶切位点的重叠序列。
引物4(seqidno.12):gagggcgtgaatgtaagcgtgacataactaattacatgattatttgtatagttcatcca,下划线序列为cyc1t的重叠序列。
从genebank基因库中查询获得终止子cyc1t片段,化学合成后扩增获得cyc1t基因片段(以引物5、引物6扩增获得)。
引物序列为:
引物5(seqidno.13):ctgggattacacatggcatggatgaactatacaaataatcatgtaattagttatgtcac,下划线序列为egfp的重叠序列。
引物6(seqidno.14):cgagctcgcaaattaaagccttcgagcg,下划线序列为saci酶切位点的重叠序列。
设计缺失启动子的基因表达盒,通过将双功能元件构建至表达盒前即可验证其终止功能是否完整。
利用引物3和引物6,以egfp和cyc1t基因片段为共同模板,通过重叠延伸pcr方法连接构建成egfp-cyc1t;
用noti和saci限制性内切酶对片段egfp-cyc1t和单拷贝质粒prs41h分别进行bamhi与noti双酶切,柱回收后,用t7连接酶将egfp-cyc1t和线性化的质粒prs41h连接,形成启动功能验证载体质粒1:prs41h-egfp-cyc1t。
将扩增获得启动子序列和验证载体1使用bamhi和noti双酶切。将酶切完成的载体和启动子序列柱回收后t7连接,形成一个完整的表达载体:prs41h-promoter-egfp-cyc1t。通过荧光蛋白的表达量确定启动子的功能及强度。核心启动子验证结果见图1。
以未做改动的空酵母cen.pk2-1c和未添加启动子序列的验证载体1酵母转化子emp-b1,作为阴性对照。dcp1、dcp2、分别为取不同随机序列的核心启动子序列。scp中的tata框类型与dcp不同,从图1可以看出不同的随机序列及不用的tata框类型可使核心启动子的强度发生变化,mutadcp2是将dcp2核心启动子序列中的tata框元件进行了突变,其荧光强度与空白值一致,说明tata框为重要的核心启动子元件。shotdcp1为随机序列截短后的dcp1核心启动序列,其强度较dcp1有所降低
上游活性序列与核心启动子序列结合后的完整启动子强度验证结果见图2。
由图2结果可以看出,在添加由两种转录因子结合位点组成的上游活性序列uas后,启动子转录水平有了明显提升,2.0uas为添加了4种转录因子结合位点的上游活性序列,启动子强度有了进一步的提升。在uas与突变核心启动子dcp2中tata序列结合后。
实施例2
终止功能的设计与验证。
在ncbi数据库中查询酿酒酵母基因组序列,通过生物信息学分析获得酿酒酵母结构基因下游的3’-utr终止子的序列信息,其中终止序列由效率元件:tatatata,定位元件:aataaa及ploy(a):tttcaaa组成。将上述元件顺序列连接成人工终止子调控元件。具体序列如下(seqidno.15):
aagacagtttatatataacgatagtaataaattattcagttgataatctcgtttcaaagcagacgtgccta。下划线序列为上述元件依次顺序连接,其余序列为随机连接序列。其中随机序列的不同也会造成终止效率的改变,本实施例仅取这一序列做讲解。
化学合成后扩增获得终止序列片段(以引物7、引物8扩增获得)。
扩增引物为:
引物7(seqidno.16):cgggatccaagacagtttatatataacgatag,下划线序列为bamhi酶切位点的重叠序列。
引物8(seqidno.17):ttttccttttgcggccgctaggcacgtctgc,下划线序列为noti酶切位点的重叠序列。
验证载体质粒2的构建:
通过从genebank查询获得酵母组成型启动子tys1p基因序列,提取酿酒酵母基因组,扩增获得tys1p基因片段(以引物9、引物10扩增获得)。
引物序列为:
引物9(seqidno.18):acgcgtcgactccttgcgcttactcgaatag<下划线序列与sali酶切位点的重叠序列。
引物10(seqidno.19):5’>gacaactccagtgaaaagttcttctcccttgctcaccattgttatcgtcaattagagta下划线序列与egfp的重叠序列。
从genebank基因库中查询获得报告基因egfp片段,化学合成后扩增获得egfp基因片段(以引物11、引物12扩增获得)。
引物序列为:
引物11(seqidno.20):cgggatcccgtatatatatataactg,下划线序列为bamhi酶切位点的重叠序列。
引物12(seqidno.21):attagcatccataaccgcatactctaattgacgataacaatggtgagcaagggagaaga,下划线序列为与启动子tys1p重叠序列。
设计缺失终止子的基因表达盒,通过将双功能元件构建至表达盒后即可验证其终止功能是否完整。
利用引物9和引物12,以tys1p和egfp基因片段为共同模板,通过重叠延伸pcr方法连接构建成tys1p-egfp;
用sali和bamhi限制性内切酶对tys1p-egfp和单拷贝质粒prs41h分别进行双酶切,柱回收后,用t7连接酶将tys1p-egfp和线性化prs41h连接,形成终止功能验证载体质粒2:prs41h-tys1p-egfp。
将扩增获得的不同终止序列和验证载体2使用bamhi和noti双酶切。将酶切完成的载体和终止序列柱回收后t7连接,形成一个完整的表达载体:prs41h-yts1p-egfp-terminator。通过荧光蛋白的表达量确定终止子的功能及强度。终止子功能验证结果见图3。
control为缺失终止子的阴性对照,t1、t2、t3为添加不同linker序列后的终止序列,t4为linker序列过长的终止序列,其余三个分别为突变效率元件、位置元件及ploy(a)后的终止序列。由图3可知,在添加不同的随机序列后对终止子的强度影响不大,但是在突变效率元件、定位元件、及ploy(a)时,可大幅降低终止子强度。当随机序列过长时,也会降低终止子强度,所以终止序列中的三个元件不宜变化且距离不能过长。
实施例3
启动终止双功能元件的构建及验证
将上述实施例中表达强度适中的启动与终止序列进行序列分析。由于终止子元件间不宜插入过长片段,所以将终止序列插入启动序列中。将达到基础转录水平的核心启动子序列置于上游活性序列后,分别将终止序列放置于启动子序列之前、上游活性序列与核心启动子序列之间。检测其转录水平使其启动和终止功能完整且互不影响。形成一种具有启动、终止双功能调控元件。具体序列如下:
化学合成后扩增获得双功能元件序列(以引物13、引物14扩增获得)。
引物序列为:
引物13(seqidno.22):cgggatccaagacagtttatatataacg,下划线序列为bamhi酶切位点的重叠序列。
引物14(seqidno.23):ttttccttttgcggccgccggtccgtagtgctcggctc,下划线序列为noti酶切位点的重叠序列。
验证载体质粒3的构建:
与实施例1相同的方法,不同的酶切位点扩增cyc1t(以引物15、引物16扩增获得)
引物15(seqidno.24):attgtgatcttccttcaaagttaggccataaacttaactcatgtaattagttatgtcac,下划线序列为mtagbfp2的重叠序列。
引物16(seqidno.25):cgagctcgcaaattaaagccttcgagc,下划线序列为saci酶切位点的重叠序列。
从genebank基因库中查询获得蓝色荧光蛋白mtagbfp2片段,化学合成后扩增获得mtagbfp2基因片段(以引物17、引物18扩增获得)。
引物序列为:
引物17(seqidno.26):ataagaatgcggccgcatggtttcaaagggtgaggaac,下划线序列为noti酶切位点的重叠序列。
引物18(seqidno.27):ggcgtgaatgtaagcgtgacataactaattacatgagttaagtttatggcctaactttg,下划线序列为cyc1t的重叠序列。
利用引物15和引物18,以mtagbfp3和cyc1t基因片段为共同模板,通过重叠延伸pcr方法连接构建成mtagbfp3-cyc1t;
用noti和saci限制性内切酶对mtagbfp2-cyc1t和验证载体1prs41h-tys1p-egfp分别进行双酶切,柱回收后,用t7连接酶将mtagbfp2-cyc1t和线性化验证载体2连接,形成双功能验证载体3:prs41h-tys1p-egfp-bamhi-noti-mtagbfp2-cyc1t。
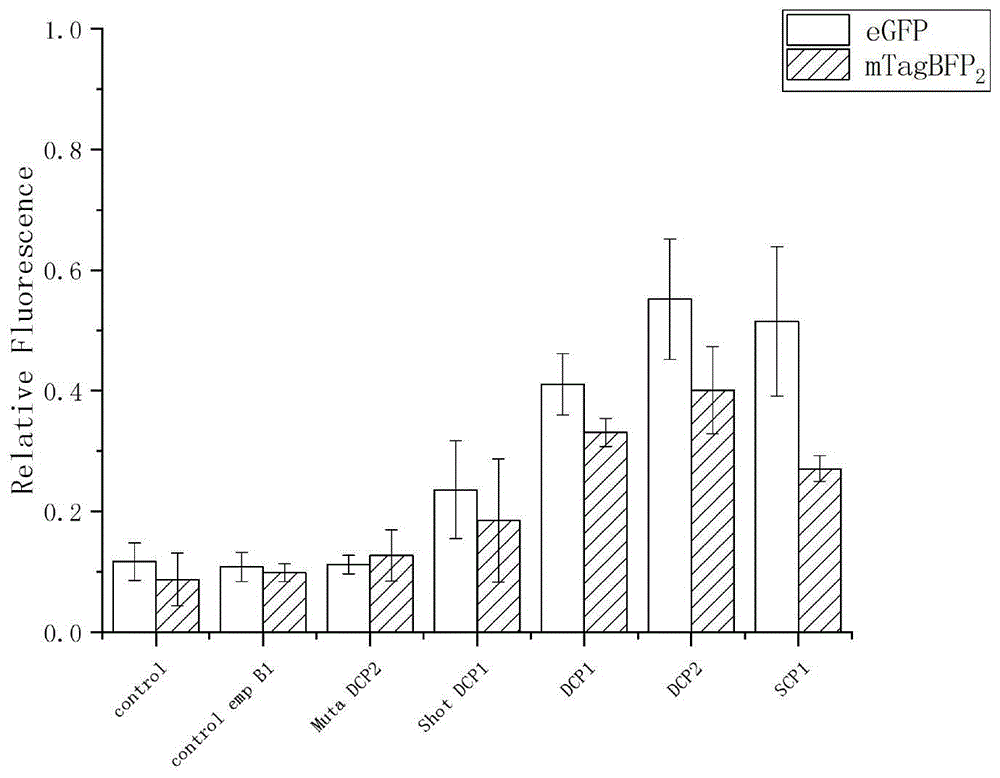
将扩增获得的双功能元件和验证载体3使用bamhi和noti双酶切。将酶切完成的载体和双功能元件柱回收后t7连接,形成一个完整的表达载体:prs41h-tys1p-egfp-双功能元件-mtagbfp2-cyc1t。通过检测两种荧光蛋白的表达量确定双功能元件的功能完整性及强度。双功能元件功能验证结果见图4。
本实施例中提及的双功能元件序列为t+2.0uas+dcp2,t为终止序列;其余上游活性序列uas、核心启动子dcp1及scp为实施例1中提及的启动子元件内容,2.0uas代表添加了两种转录因子的上游活性序列,onlyt+uas代表缺失核心启动序列的双功能元件。由图4可以看出双功能元件的功能完整,可以正常终止前一个转录单元中的绿色荧光蛋白,也可以正常启动第二个转录单元中的蓝色荧光蛋白。通过缺失核心启动序列,第二转录单元表达量明显下降,说明核心启动序列对启动功能影响很大。通过改变双功能序列中的元件顺序可以导致两个转录单元的表达量发生变化。但是当终止序列置于启动子序列之后对启动功能有很大影响,所以仅理性结合方式仅选用第一种:终止序列置于启动序列之前,以及第二种:终止序列置于上游活性序列与核心启动子序列之间,这两种结合方式。
将上述实施例中连接形成的三种功能验证载体1prs41h-egfp-cyc1t、验证载体2prs41h-tys1p-egfp、验证载体3prs41h-tys1p-egfp-bamhi-noti-mtagbfp2-cyc1t分别转化大肠杆菌感受态细胞,涂在含有amp抗性的筛选平板上,经过菌落pcr和测序验证获得含有验证载体的三种大肠杆菌工程菌。
实施例4
双功能元件的构建及双功能元件库的建立及应用
分别挑取含有实施例3的验证载体1、2、3的大肠杆菌单菌落,接种于含有2%氯化钠、2%蛋白胨和1%酵母粉的液体培养基中,37℃、150rpm震荡过夜培养,提取质粒,三个验证载体均使用bamhi和noti双酶切,使其线性化,用于双功能元件连接。将扩增获得的双功能元件序列使用bamhi和noti双酶切。将酶切完成的载体和双功能元件柱回收后t7连接,形成三个完整的表达载体:tys1p-egfp-双功能元件、双功能元件-egfp-cyc1t、tys1p-egfp-双功能元件-mtagbfp2-cyc1t。
将三种表达载体转化至大肠杆菌感受态细胞中,后涂布于amp抗性平板,过夜培养后挑取单菌落进行菌落pcr验证,取阳性结果的菌株接种于含有2%氯化钠、2%蛋白胨和1%酵母粉的液体培养基中,37℃、150rpm震荡过夜培养,提取质粒。将构建完成的三个表达质粒用醋酸锂转化法转化到酿酒酵母cen.pk2-1c细胞中,获得酿酒酵母转化子。
选择不同结构的双功能元件进行扩增,按照上述步骤,可获得含有不同双功能元件表达载体和酿酒酵母转化子。
选择双功能元件库中的不同双功能元件表达载体的酿酒酵母转化子接种于含有2%葡萄糖、2%蛋白胨和1%酵母粉的液体培养基中,30℃、220rpm震荡培养36小时,利用流式细胞仪检测细胞内egfp和mtagbfp2的荧光强度,以酿酒酵母cen.pk2-1c及空验证载体转化子的荧光强度为空白,表达盒tys1p-egfp-cyc1t的荧光强度为参照,发现本发明构建的双功能元件可以正常行使启动和终止的功能,验证不同强度的双功能元件后形成双功能元件库。
在通过改变元件的排列、不同元件间的连接序列及不同元件的选用后,可以得到一系列不同强度的双功能元件库
元件库中代号的具体结合方式见表1。
表1元件库中代号的具体结合方式
不同强度的双功能元件表达情况见图5。
实施例5:双功能元件在番茄红素途径构建中的应用。
本案例选取番茄红素异源合成途径为验证对象,选择番茄红素合成途径中的2个关键合成酶:欧文氏菌的八氢番茄红素合成酶crtb(genbank:kc954270.1)和来自红发夫酵母的八氢番茄红素去饱和酶crti(genbank:ay177424.1),在体外构建质粒表达盒。分别使用天然启动子和终止子与双功能元件做对比,天然启动子选择cyc1p,tys1p;终止子选择adh1t,pdalt。以oe-pcr和gibsonassembly两种组装方法同时组装成含有prs41h-(cyc1p-crtb-adh1t)-(tys1p-crti-adalt)的完整质粒。
通过从genebank查询获得酵母启动子cyc1p基因序列,化学合成后,扩增获得cyc1p基因片段(以引物19、引物20扩增获得)。
引物19(seqidno.28):ccgggccccccctcgagggcatgcatgtgctctgtatgtatataaaac,下划线序列为载体质粒prs41h的重叠序列。
引物20(seqidno.29):gttcaacaaagatgggttgttcattattaatttagtgtgtgtatttgtgtttgtgtgtc,下划线序列为crtb的重叠序列。
通过从genebank查询获得欧文氏菌的八氢番茄红素合成酶crtb(genbank:kc954270.1)基因序列,化学合成后,扩增获得crtb基因片段(以引物21、引物22扩增获得)。
引物21(seqidno.30):gacacacaaacacaaatacacacactaaattaataatgaacaacccatctttgttgaac,下划线序列为cyc1p的重叠序列。
引物22(seqidno.31):ctttaaaatttgtatacacttattttttttataactttacaatggtctttgccacaagt,下划线序列为adh1t的重叠序列。
通过从genebank查询获得酵母终止子adh1t基因序列,提取酿酒酵母基因组,扩增获得adh1t基因片段(以引物23、引物24扩增获得)
引物23(seqidno.32):acttgtggcaaagaccattgtaaagttataaaaaaaataagtgtatacaaattttaaag,下划线序列为crtb的重叠序列。
引物24(seqidno.33):cctattcgagtaagcgcaaggatcggcatgccggtagagg,下划线序列为tys1p的重叠序列。
通过从genebank查询获得酵母终止子tys1p基因序列,提取酿酒酵母基因组,扩增获得tys1p基因片段(以引物25、引物26扩增获得)。
引物25(seqidno.34):cctctaccggcatgccgatccttgcgcttactcgaatagg,下划线序列为adh1t的重叠序列。
引物26(seqidno.35):gtcttggtcttgttccttacccattgttatcgtcaattagagtatgcgg,下划线序列为crti的重叠序列。
通过从genebank查询获得红发夫酵母的八氢番茄红素去饱和酶crti(genbank:ay177424.1)基因序列,化学合成后,扩增获得crti基因片段(以引物27、引物28扩增获得)
引物27(seqidno.36):ccgcatactctaattgacgataacaatgggtaaggaacaagaccaagac,下划线序列为tys1p的重叠序列。
引物28(seqidno.37):ccttacgatttaattaatccctttagaaagccaaaacaccaacagatc,下划线序列为adalt的重叠序列。
通过从genebank查询获得酵母终止adalt基因序列,提取酿酒酵母基因组,扩增获得adalt基因片段(以引物29、引物30扩增获得)。
引物29(seqidno.38):gatctgttggtgttttggctttctaaagggattaattaaatcgtaagg,下划线序列为crti的重叠序列。
引物30(seqidno.39:ggcggccgctctagaactagtgcccaccagacttcaatttttg,下划线序列为的prs41h重叠序列。
以prs41h空载体为模板,扩增获得线性化的prs41h基因片段(以引物31、引物32扩增获得)。
引物31(seqidno.40:caaaaattgaagtctggtgggcactagttctagagcggccgcc,下划线序列为adalt的重叠序列。
引物32(seqidno.41:gttttatatacatacagagcacatgcatgccctcgagggggggcccgg,下划线序列为cyc1p的重叠序列。
以上述引物获得的基因片段,同时使用一步连接,构建成含有prs41h-cyc1p-crtb-adh1t-tys1p-crti-adalt的完整质粒。
以双功能元件做对照,选用双功能元件库中的u7、u8、u9替代所用的启动子cyc1p、tys1p,终止子adh1t、pdalt。
以相同的实验条件,操作手法,不同的组装方法,并作3组平行实验。做出以下对比(表2)对比后发现使用双功能元件简化构建步骤后,使用不同dna组装方法其连接效率约有18%的提升。
表2双功能元件用于途径组装时的效果
以上所述仅是本发明的优选实施方式,并非对本发明作任何形式上的限制。应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>石河子大学
<120>一种具有启动、终止双功能调控元件的构建方法、双功能元件库和应用
<160>41
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>1
gatcgtagccccggatttac20
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
gatcgtacacctggacatac20
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
gatcgtagagcttccactac20
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gatcgtagccccacaaatac20
<210>5
<211>11
<212>dna
<213>人工序列(artificialsequence)
<400>5
cgagccgagca11
<210>6
<211>304
<212>dna
<213>人工序列(artificialsequence)
<400>6
aagacagtttatatataacgatagtaataaattattcagttgataatctcgtttcaaagc60
agacgtgcctaagtgataatcgatcgtagccccggatttacgtaagttctggatcgtaca120
cctggacatacagattgtgatggcaatctcgattacactgatcgtagagcttccactact180
aaatcggtgatcgtagccccacaaataccgtgatactgtattgtattttcacaggacgcc240
tatataaaactcttgttttcttcttttctcttaatattcttttcgagccgagcactacgg300
accg304
<210>7
<211>147
<212>dna
<213>人工序列(artificialsequence)
<400>7
agtgataatcgatcgtagccccggatttacgtaagttctggatcgtacacctggacatac60
agattgtgatggcaatctcgattacactgatcgtagagcttccactactaaatcggtgat120
cgtagccccacaaataccgtgatactg147
<210>8
<211>86
<212>dna
<213>人工序列(artificialsequence)
<400>8
tattgtattttcacaggacgcctatataaaactcttgttttcttcttttctcttaatatt60
cttttcgagccgagcactacggaccg86
<210>9
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>9
cgggatccagtgataatcgatcg23
<210>10
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>10
ttttccttttgcggccgccggtccgtagtgctcggc36
<210>11
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>11
ataagaatgcggccgcatggtgagcaagggagaagaac38
<210>12
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>12
gagggcgtgaatgtaagcgtgacataactaattacatgattatttgtatagttcatcca59
<210>13
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>13
ctgggattacacatggcatggatgaactatacaaataatcatgtaattagttatgtcac59
<210>14
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>14
cgagctcgcaaattaaagccttcgagcg28
<210>15
<211>71
<212>dna
<213>人工序列(artificialsequence)
<400>15
aagacagtttatatataacgatagtaataaattattcagttgataatctcgtttcaaagc60
agacgtgccta71
<210>16
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>16
cgggatccaagacagtttatatataacgatag32
<210>17
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>17
ttttccttttgcggccgctaggcacgtctgc31
<210>18
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>18
acgcgtcgactccttgcgcttactcgaatag31
<210>19
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>19
gacaactccagtgaaaagttcttctcccttgctcaccattgttatcgtcaattagagta59
<210>20
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>20
cgggatcccgtatatatatataactg26
<210>21
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>21
attagcatccataaccgcatactctaattgacgataacaatggtgagcaagggagaaga59
<210>22
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>22
cgggatccaagacagtttatatataacg28
<210>23
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>23
ttttccttttgcggccgccggtccgtagtgctcggctc38
<210>24
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>24
attgtgatcttccttcaaagttaggccataaacttaactcatgtaattagttatgtcac59
<210>25
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>25
cgagctcgcaaattaaagccttcgagc27
<210>26
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>26
ataagaatgcggccgcatggtttcaaagggtgaggaac38
<210>27
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>27
ggcgtgaatgtaagcgtgacataactaattacatgagttaagtttatggcctaactttg59
<210>28
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>28
ccgggccccccctcgagggcatgcatgtgctctgtatgtatataaaac48
<210>29
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>29
gttcaacaaagatgggttgttcattattaatttagtgtgtgtatttgtgtttgtgtgtc59
<210>30
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>30
gacacacaaacacaaatacacacactaaattaataatgaacaacccatctttgttgaac59
<210>31
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>31
ctttaaaatttgtatacacttattttttttataactttacaatggtctttgccacaagt59
<210>32
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>32
acttgtggcaaagaccattgtaaagttataaaaaaaataagtgtatacaaattttaaag59
<210>33
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>33
cctattcgagtaagcgcaaggatcggcatgccggtagagg40
<210>34
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>34
cctctaccggcatgccgatccttgcgcttactcgaatagg40
<210>35
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>35
gtcttggtcttgttccttacccattgttatcgtcaattagagtatgcgg49
<210>36
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>36
ccgcatactctaattgacgataacaatgggtaaggaacaagaccaagac49
<210>37
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>37
ccttacgatttaattaatccctttagaaagccaaaacaccaacagatc48
<210>38
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>38
gatctgttggtgttttggctttctaaagggattaattaaatcgtaagg48
<210>39
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>39
ggcggccgctctagaactagtgcccaccagacttcaatttttg43
<210>40
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>40
caaaaattgaagtctggtgggcactagttctagagcggccgcc43
<210>41
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>41
gttttatatacatacagagcacatgcatgccctcgagggggggcccgg48
- 还没有人留言评论。精彩留言会获得点赞!